Séquençage des protéines par spectrométrie de masse
Outils
Général
Imprimer / exporter

Cet article doit être recyclé ().
Une réorganisation et une clarification du contenu paraissent nécessaires. Améliorez-le, discutez des points à améliorer ou précisez les sections à recycler en utilisant {{section à recycler}}.
Le séquençage des protéines par spectrométrie de masse est une technique qui permet le séquençage des protéines par spectrométrie de masse surtout en tandem MS/MS. Elle permet de déterminer la séquence des peptides sur quelques dizaines d'acides aminés. Couplé avec des techniques de digestion (enzymatiques ou chimiques) et avec des techniques de séparation (chromatographie en phase liquide à haute performance par exemple), il est possible de caractériser une protéine.
La fragmentation est le processus permettant de séparer chaque acide aminé d'un peptide. Pour des raisons de stabilité des liaisons, les séquences polypeptidiques se clivent préférentiellement au niveau des liaisons peptidiques. Il existe cependant des mécanismes complexes qui produisent d'autres types ruptures de liaison, ce qui peut rendre difficile la lecture des spectres de masses et la détermination de la séquence.
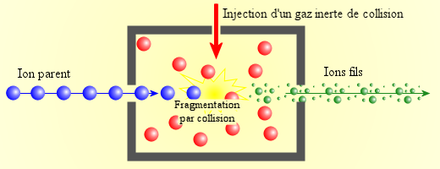
Pour fragmenter les peptides, on utilise soit plusieurs analyseurs couplés en série (spectromètre de type triple quadripoles QqQ ou de type quadripole-temps de vol Q-TOF) ou un analyseur capable de réaliser plusieurs analyses en série (spectromètre de masse de type trappe ionique IT ou de type résonance cyclotronique FT-ICR).
Il existe plusieurs mode d'analyse en spectrométrie de masse en tandem :
Les fragmentations peuvent être classées en fonction de leurs types :
En fonction de l'énergie interne des ions, les ions se fragmentent à différents temps de vie :

M = M(n AA) + M(H) + M(OH)
| Code à 1 lettre | Code à 3 lettres | Acide aminé | Masses | Acides aminés modifiés |
|---|---|---|---|---|
| A | Ala | Alanyl | 71 | 113 (Acétylation) 297 (Biotinylation) |
| C | Cys | Cystyl | 103 | 160 (Carbamidométhylation) 161(Carboxyméthyl) 174 (Propionamidation) 202 (NIPCAM) 208 (S-pyridyléthylation) |
| D | Asp | Aspartyl | 115 | 129 (Méthylestérification) 137 (Sodiation) |
| E | Glu | Glutamyl | 129 | 111 (Pyro-glu) 143 (Méthylestérification) 151 (Sodiation) |
| F | Phe | Phénylalanyl | 147 | - |
| G | Gly | Glycyl | 57 | - |
| H | His | Histidyl | 137 | 153 (Oxydation) |
| I | Ile | Isoleucyl | 113 | - |
| K | Lys | Lysyl | 128 | 255 (SMA) |
| L | Leu | Leucyl | 113 | - |
| M | Met | Méthionyl | 131 | 147 (Oxydation) 163 (Sulfonation) |
| N | Asn | Asparagyl | 114 | - |
| P | Pro | Prolyl | 97 | - |
| Q | Gln | Glutamyl | 128 | 111 (Pyro-glu en N-term) |
| R | Arg | Arginyl | 156 | - |
| S | Ser | Séryl | 87 | 167 (Phosphorylation) |
| T | Thr | Thréonyl | 101 | 181 (Phosphorylation) |
| V | Val | Valyl | 99 | - |
| W | Trp | Tryptophanyl | 186 | 206 (Oxydation) |
| Y | Tyr | Tyrosyl | 163 | 243 (Phosphorylation) |
| Modification | Masse |
|---|---|
| Acétylation | + 42 Da |
| Biotinylation | + 226 Da |
| Carbamylation | + 43 Da |
| Formylation | + 28 Da |
| SMA | + 127 Da |
| Modification | Masse |
|---|---|
| Méthylestérification | + 14 Da |
| Sodiation | + 22 Da |
La fragmentation de la série a produit des ions aldimines :

M = M(n AA) + M(H) - M(CO) = M(n AA) - 27
La série a* dérive de la série a par la perte d'un groupe NH3 :
M = a - M(NH3) = a - 17
La série a0 dérive de la série a par la perte d'un groupe H2O :
M = a - M(H2O) = a - 18
La fragmentation de la série b produit des ions acylium :

M = M(n AA) + M(H) = M(n AA) + 1
La série b* dérive de la série b par la perte d'un groupe NH3 :
M = b - M(NH3) = b - 17
La série b0 dérive de la série b par la perte d'un groupe H2O :
M = b - M(H2O) = b - 18
Le fragment peptidique de plus faible poids moléculaire de la série b correspond à l'extrémité N terminal du peptide parent. Il correspond à la formule :
RNH2-CH-CO
et il donne un pic avec un rapport m/z :
m/z = M(H) + M(aa) = M(aa) + 1
Le fragment peptidique de plus haut poids moléculaire de la série b correspond à l'extrémité C terminal du peptide parent [M+H]+. Il correspond à la formule :
RNH2-...-(CH)-...-CO
La différence de masse entre ce fragment et le peptide parent correspond donc au dernier acide aminé aan :
m/z = M(ion parent M[M+H]+) - M(fragment peptidique) = (M(H) + ΣM(aa) + M(aan + M(OH) + M(H+)) - (M(H) + ΣM(aa)) = M(aan) + 18
En particulier, dans le cas d'une digestion trypsique, l'acide aminé C terminal correspond à une lysine ou une arginine. En fonction de cet acide aminé, on observe un pic qui apparait aux différence suivante par rapport au rapport m/z de l'ion parent [M+H]+ :
m/z = 146 pour la lysine
et
m/z = 174 pour l'arginine
La fragmentation de la série c produit des ions amino :
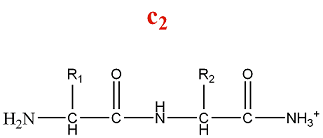
M = M(n AA) + M(H) + M(NH3) = M(n AA) + 18
La fragmentation de la série x produit des ions acylium :
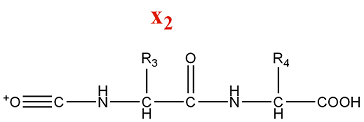
M = M(n AA) + M(CO) + M(OH) = M(n AA) + 45
La fragmentation de la série y produit des ions amino :

M = M(n AA) + M(2H) + M(OH) = M(n AA) + 19
La série y* dérive de la série y par la perte d'un groupe NH3 :
M = y - M(NH3) = y - 17
La série y0 dérive de la série y par la perte d'un groupe H2O :
M = y - M(H2O) = y - 18
Le fragment peptidique de plus faible poids moléculaire de la série y correspond à l'extrémité C terminal du peptide parent. Il correspond à la formule :
RNH3+-CH-COO
et il donne un pic avec un rapport m/z :
m/z = 2M(H) + M(aa) + M(OH) = M(aa) + 19
En particulier, dans le cas d'une digestion trypsique, l'acide aminé C terminal correspond à une lysine ou une arginine. En fonction de cet acide aminé, on observe un pic aux valeurs :
m/z = 147 pour la lysine
et
m/z = 175 pour l'arginine
Le fragment peptidique de plus haut poids moléculaire de la série y correspond à l'extrémité N terminal du peptide parent. Il correspond à la formule :
RNH3+-...-(CH)-...-COO
La différence de masse entre ce fragment et l'ion parent [M+H]+ correspond donc au premier acide aminé aa1 :
m/z = M(ion parent M[M+H]+) - M(fragment peptidique) = (M(H) + ΣM(aa) + M(aa1 + M(OH) + M(H+)) - (2M(H) + ΣM(aa) + M(OH)) = M(aa1)
M = M(n AA) - M(NH) + M(OH) = M(n AA) + 2
Ces ions correspondent au clivage de deux liaisons peptidiques. Ces clivages correspondent en général à des fragmentations de types b et y et forment des ions amino-acylium :

M = M(n AA) + M(2H) = M(n AA) + 2
Parfois, les clivages correspondent à des fragmentations de types a et y et produisent des ions amino-iminium :
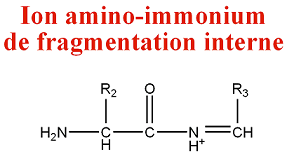
M = M(n AA) + M(H) - M(CO) = M(n AA) - 27
Lorsqu'une fragmentation de type a est voisine d'une fragmentation de type y, il se forme un ion iminium :

M = M(AA) + M(H) - M(CO) = M(AA) - 27
Les ions immoniums apparaissent dans un spectre MS/MS à des masses inférieurs à 200 Da. Ils permettent de connaître la composition en acide aminé du peptide fragmenté.
| Acide aminé | Code à 3 lettres | Code à 1 lettres | Masse | Ion immonium dérivé |
|---|---|---|---|---|
| Acide aspartique | Asp | D | 88 | 70 |
| Acide glutamique | Glu | E | 102 | - |
| Alanine | Ala | A | 44 | - |
| Arginine | Arg | R | 129 | 59, 70, 73, 87, 100, 112 |
| Asparagine | Asn | N | 87 | 70 |
| Cystéine | Cys | C | 76 | - |
| Glutamine | Gln | Q | 101 | 56, 84, 129 |
| Glycine | Gly | G | 30 | - |
| Histidine | His | H | 110 | 82, 121, 123, 138, 166 |
| Isoleucine | Iso | I | 86 | 44, 72 |
| Leucine | Leu | L | 86 | 44, 72 |
| Lysine | Lys | K | 101 | 70, 84, 112, 129 |
| Méthionine | Met | M | 104 | 61 |
| Phénylalanine | Phe | F | 120 | 91 |
| Proline | Pro | P | 70 | - |
| Sérine | Ser | S | 60 | - |
| Thréonine | Thr | T | 74 | - |
| Tryptophane | Trp | W | 159 | 77, 117, 130, 132, 170, 171 |
| Tyrosine | Tyr | Y | 136 | 91, 107 |
| Valine | Val | V | 72 | 41, 55, 69 |
Un ion de la série d est formé par la perte d'une partie de la chaîne latérale de l'acide aminé C terminal d'un ion de la série a.
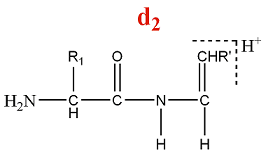
M = M(AA) + M(H) - M(CO) - M(Rperdu) = M(AA) - 27 - M(Rperdu)
Un ion de la série v est formé par la perte de la chaîne latérale de l'acide aminé N terminal d'un ion de la série y.
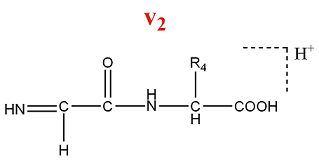
M = M(AA) + M(OH) - M(Rperdu) = M(AA) + 17 - M(Rperdu)
Un ion de la série w est formé par la perte d'une partie de la chaîne latérale de l'acide aminé N terminal d'un ion de la série z.
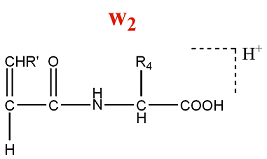
M = M(AA) + M(OH) - M(NH) - M(Rperdu) = M(AA) + 2 - M(Rperdu)
| Spectroscopique | |
|---|---|
| Chimique | |
| Informatique | |
| Séquençage | |