RNAポリメラーゼ
RNAポリメラーゼ[注釈 1] (RNA polymerase) とは、リボヌクレオチドを重合させてRNAを合成する酵素(RNA合成酵素)。
DNAの鋳型鎖(一本鎖)の塩基配列を読み取って相補的なRNAを合成する反応(転写)を触媒する中心となる酵素をDNA依存性RNAポリメラーゼという(単に「RNAポリメラーゼ」とも呼ぶ)。真核生物では、DNAを鋳型にしてmRNAやsnRNA遺伝子の多くを転写するRNAポリメラーゼIIがよく知られる。このほかに35S rRNA前駆体を転写するRNAポリメラーゼI、tRNAとU6 snRNA、5S rRNA前駆体等を転写するRNAポリメラーゼIIIなどがあり、この三種はDNA依存性RNAポリメラーゼと呼ばれる。
また、RNAを鋳型にRNAを合成するRNA依存性RNAポリメラーゼもあり、多くのRNAウイルスで重要な機能を果たす以外に、microRNAの増幅過程にも利用される。
鋳型を必要としない物もあり、初めて発見されたRNA ポリメラーゼであるポリヌクレオチドホスホリラーゼ(ポリヌクレオチドフォスフォリレース、ポリニュークリオタイドフォスフォリレース)もそのひとつとしてあげられる。この酵素は実際には細菌の細胞内でヌクレアーゼとして働くが、試験管内ではRNAを合成することができる。これを利用して一種類のヌクレオチドからなるRNAを合成し、それから翻訳されるタンパク質を調べることで初めて遺伝暗号の決定が行われた。真核生物のもつpoly(A)ポリメラーゼも同様に鋳型を必要とせず、Pol II転写産物の3'末端にpoly(A)鎖を付加することで転写後の遺伝子発現制御機構の一端を担っている。
真核生物の転写装置(RNAポリメラーゼ)は、Pol I、Pol II、Pol IIIの3種がある。それぞれ10種類以上ものサブユニットから構成される(基本的には12種)。また、古細菌のRNAポリメラーゼもサブユニット数が多く、9-14種のサブユニットから構成されている。ユーリ古細菌ではいくつかのサブユニットが省かれているが、一部のクレン古細菌には真核生物の12種類のサブユニットが全て保存されており、真核生物の持つ3種のRNAポリメラーゼの祖先型と考えられている。古細菌のRNAポリメラーゼは、Aサブユニットが2つに分かれている特徴がある。
一方で、真正細菌のRNAポリメラーゼは全体的に真核生物や古細菌のものより単純な構成である。ααββ'ωの4種5サブユニットからなるコアエンザイムに、σが会合したホロエンザイムと呼ばれる形態で正常なプロモーターを認識する。シグマ因子は遺伝子上流のプロモーター配列を認識して転写を開始する役割を担っている。
真正細菌のRNAポリメラーゼサブユニット
大腸菌のRNAポリメラーゼホロ酵素RNA polymerase holoenzyme は2分子のα (α1, 2) および1分子ずつのβ、β’、σ、ω サブユニットを含む[1]。σサブユニット以外だけでも複合体を形成し、これをRNAポリメラーゼコア酵素 (RNA polymerase core enzyme, コアポリメラーゼ (core polymerase))と呼ぶ。コア酵素は実際にRNAを合成する部位で、σサブユニット(σ因子)はコア酵素を特定の遺伝子に導き、ホロ酵素の特異性 specificity (σはギリシャ文字のs) を担うといえる[2]。
それぞれの項で各サブユニットを紹介する。
αサブユニット
RNAポリメラーゼホロ酵素において2つ存在するαサブユニットは、開始段階ではプロモーターのUPエレメントの認識を担う。一方、伸長段階になるとコア酵素の会合を含む様々な活性を示す。
リチャード・グルース (Richard Grouse) らはα235 (C末端の94アミノ酸が欠損。正常なαサブユニットのアミノ酸数は329であるため、94アミノ酸を失い235) およびR265C (N末端から265番目のアルギニンをシステインに置換) という2つのαサブユニット変異体について実験を行った。これにより、RNAポリメラーゼホロ酵素がUPエレメントを認識しないことが明らかにされた[3]。また、グルースとリチャード・エブライト (Richard Ebright) らはタンパク質限定分解法を用いて、αサブユニットのN末端およびC末端がそれぞれ独立してα-NTD (amino terminal domain of the α subunit) およびα-CTD (carboxyl terminal domain of the α subunit) というドメインを形成することを突き止めた[4]。実験に用いられた生物は大腸菌である。N末端ドメインは8〜241付近を含む28 kD、C末端ドメインは249〜329(末端)付近を含む8 kDである。グルースとエブライトらはまた、両者が明確な構造 (モチーフ) をとらない、少なくとも239〜251の13アミノ酸による連結鎖でつながっていることも発見した[5]。
このことから、α-CTDの機能について一つの仮説が考えられる。RNAコア酵素においてほかのタンパク質と相互作用するのはα-NTDであり、αCTDは連結鎖の先でコア酵素から離れている。しかし、UPエレメントに対して強力に結合し、DNAとホロ酵素とのつながりをさらに強固に補う[5][6]。後述するRF複合体の立体構造解析から、2つあるUPエレメントのうち-40付近のものはα1が、-60付近のものはα2が連結することが示されている。
βサブユニット
β'サブユニットとともに転写産物の伸長を担う。どちらもDNAとの結合部位を持つが、βサブユニットのそれはN末端[注釈 2]近くのMet30〜Met102の領域である[7]。静電相互作用で弱く結合する。エフゲニー・ナドラー (Evgeny Nudler) の1996年の実験によると、DNAの-6〜+1が結合標的であり、転写中この部位は融解している[8]。DNAとの接続で中心になるのは別のβ'サブユニットの結合部位であるが、βサブユニットのそれはその上流に位置する。このため、上流へと吐き出される転写産物が鋳型鎖との結合を脅かしたとしても、RNAポリメラーゼの活性に大きな影響はない。また、ナドラーの別の実験によると、βサブユニットはβ’の結合にも関わるようである[7]。
ホロ酵素の活性部位を構成するタンパク質の一つであり、補因子であるMg+と結合する3つのアスパラギン酸を持つ。
βサブユニットは微生物に対する代表的な抗生物質であるリファンピシンとストレプトリジギンの直接的な作用標的である[9]。したがってこの2つの抗生物質は転写の伸長を阻害する。ただし、ストレプトリジギンは開始段階に効果があるとされている。これは、開始段階にも10ntのRNA(アボーティブ転写産物)を合成する過程[注釈 3]があり、これを阻害するためである[9]。
β'サブユニット
β'サブユニットは、転写の開始段階においてRNAポリメラーゼホロ酵素が-11〜+1位を巻き戻すことを助ける[9]。この巻き戻しはいわゆる開放型複合体の形成[注釈 4]であるが、その際に非鋳型鎖[注釈 5]の-10領域中にRNAポリメラーゼの結合が必要である。キャロル・グロス (Carol Gross) らの研究によると、結合はβ'の262〜309のアミノ酸領域が促す[3]。
伸長段階においてはRNAポリメラーゼホロ酵素のDNA結合を担う。すなわち、C末端[注釈 2]近くのMet1230〜Met1273で+2〜+11の領域に強く疎水性相互作用する[8]。このDNA領域はβサブユニットとの結合部位と異なり、転写中は二重らせんのままである。
σサブユニット
| σ70 領域1.1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| 略号 | Sigma70_r1_1 | ||||||||
| Pfam | PF03979 | ||||||||
| InterPro | IPR007127 | ||||||||
| |||||||||
| σ70 領域1.2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Thermus aquaticus のRNAポリメラーゼにおける、領域1.2から3.1までのσ因子断片の結晶構造 | |||||||||
| 識別子 | |||||||||
| 略号 | Sigma70_r1_2 | ||||||||
| Pfam | PF00140 | ||||||||
| InterPro | IPR009042 | ||||||||
| PROSITE | PDOC00592 | ||||||||
| SCOP | 1sig | ||||||||
| SUPERFAMILY | 1sig | ||||||||
| |||||||||
| σ70 領域2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Escherichia coli のRNAポリメラーゼにおけるσ70断片の結晶構造 | |||||||||
| 識別子 | |||||||||
| 略号 | Sigma70_r2 | ||||||||
| Pfam | PF04542 | ||||||||
| Pfam clan | CL0123 | ||||||||
| InterPro | IPR007627 | ||||||||
| PROSITE | PDOC00592 | ||||||||
| SCOP | 1sig | ||||||||
| SUPERFAMILY | 1sig | ||||||||
| |||||||||
| σ70 領域3 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| 略号 | Sigma70_r3 | ||||||||
| Pfam | PF04539 | ||||||||
| Pfam clan | CL0123 | ||||||||
| InterPro | IPR007624 | ||||||||
| SCOP | 1ku2 | ||||||||
| SUPERFAMILY | 1ku2 | ||||||||
| |||||||||
| σ70 領域4 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Thermotoga maritima のσ70における領域4の溶液中構造 | |||||||||
| 識別子 | |||||||||
| 略号 | Sigma70_r4 | ||||||||
| Pfam | PF04545 | ||||||||
| Pfam clan | CL0123 | ||||||||
| InterPro | IPR007630 | ||||||||
| SCOP | 1or7 | ||||||||
| SUPERFAMILY | 1or7 | ||||||||
| |||||||||
| σ70 領域4.2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
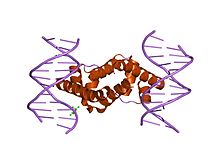 -35ボックスに結合した、Escherichia coli のσ70における領域4 | |||||||||
| 識別子 | |||||||||
| 略号 | Sigma70_r4_2 | ||||||||
| Pfam | PF08281 | ||||||||
| Pfam clan | CL0123 | ||||||||
| InterPro | IPR013249 | ||||||||
| SCOP | 1or7 | ||||||||
| SUPERFAMILY | 1or7 | ||||||||
| |||||||||
特に転写開始段階[注釈 3]で活躍するようである。σ因子があるとRNAポリメラーゼは不特定のDNA部位(緩い結合部位、loose binding site)に弱く結合する。滑って移動し、プロモーターに出会うかそのまま遊離する。これにより、RNAポリメラーゼによる転写を行う遺伝子の発見は加速される。速度定数にして1010 M-1 s-1で、滑らずにDNAへ無差別に結合と解離を繰り返す場合の100倍である[10][11]。結合した時の安定性でいえば、解離までの半減期は約60分と長い[11]。σ因子がなければ1秒以下である[11]。σサブユニットはまたRNAポリメラーゼとプロモーターを、半減期が数時間になるほど強固に結合させる。ホロ酵素とプロモーターの会合定数はほかの配列と比較して平均約107倍であり、コア酵素の平均1000倍にもなる[11]。プロモーターによって結合定数は106〜1012と幅広く、rRNAのような約1秒に1回からlacI 遺伝子のような約30分に1回という転写頻度の違いを生み出す[12]。それだけではなく、伸長段階への移行に必要なDNAの巻き戻しも担う[注釈 4][13]。
伸長段階に移行するとき、RNAポリメラーゼは構造を変えるが、このときσ因子の結合は極端に弱くなる。トラバーズ (Travers) とバージェス (Burgess) の研究によると、σ因子が伸長を促進することはない[14]。二人の1969年の論文では、離れたσ因子は別のコア酵素と結合し、なおかつそれはDNAの正常な転写を行うことが証明された[14]。このことから、σ因子は再利用されると考えられる。σサイクルという循環の中では当初、伸長前に必ず離れるものと考えられていたが、現在では結合が弱くなるだけという説が有力である[15]。実際、伸長段階に至ったホロ酵素の70%はσ因子を保有したままである[13]。すなわち、σ因子は通常伸長が止まったときに、別のコア酵素に利用されるため離れる。
特別な遺伝子を専任するσ因子もある。あらゆる真正細菌は、成長機能に関する遺伝子(通常の増殖に必要な遺伝子)を転写する主要σ因子 (primary σ factor, σA) を持つ。例えば、大腸菌ではσ70であり、枯草菌 ではσ43である[16]。それぞれ70 kDと43 kDで、右上の番号は分子量に由来する。ほかにも、熱ショック遺伝子や胞子形成遺伝子なども特別なσ因子が担当する[16]。多くの種類があるのは、環境条件によって適切な遺伝子群を発現するためで、この使い分けは特に枯草菌を用いた研究によって明らかとなった。普段はσ43が転写制御に当たっているが、栄養状態が悪くなった場合などには他のσ因子(σHなど)が発現し、胞子形成の準備を始める。その後母細胞ではE、Kと変化し、胞子ではF、Gが使用される。
σ因子の領域
あらゆる真正細菌におけるσ因子のアミノ酸配列は領域1から4に分類できる。ハーマン (Helmann) とチェンバーリン (Chamberlin) は各領域の機能を以下のように提唱した[17]。
領域1は主要σ因子にしか存在しない。σ因子がRNAポリメラーゼを伴わずにプロモーターと結合することを阻害する。このため、DNAと結合するためにはRNAポリメラーゼコア酵素と結合して、後述する領域2.4と4.2のドメインを露出させなければならない。σ因子単独の結合はコア酵素とプロモーター間の結合の阻害につながるためこの機能は重要である。
領域2は全てのσ因子に存在し、あらゆる生物で最も共通性が高い[18]。さらに領域2.1から2.4に分類される。特に重要なのは領域2.4で、これは-10ボックスに特異的に強く結合する。DNAとの結合に最適なαヘリックスを形成すると予測されるアミノ酸配列を含んでいるが、実際に-10ボックスを認識することはリチャード・ロジック (Richard Losick) が代償変異の実験で証明した[18]。
領域3はコア酵素とDNA両方の結合に関与する。領域3と4をつなげる連結鎖は、ほとんどの転写で最初に合成されるアデニンとの特異的な結合に関わり[19]、また、RNA出口通路を塞ぐ (#真正細菌のホロ酵素-DNA複合体の(3)で詳述)。合成されたばかりのアデニンはDNAとの2本の弱い水素結合でしか支えられておらず、ホロ酵素との特異的な結合が必要である。連結鎖を欠いたホロ酵素を用いた実験では、最初の2つのリボヌクレオチドの一方、または両方が通常よりはるかに高濃度でなければ転写が始まらないことが確認された[19]。
領域4は4.1と4.2に分けられ、ホロ酵素のプロモーター認識において重要と考えられている。領域4.2はヘリックスターンヘリックスというDNA結合ドメインを含み、-35ボックスに強く結合する。
真正細菌の伸長複合体
伸長段階を実行するDNAポリメラーゼを中心とした複合体の立体構造についての研究は、1999年にセス・ダースト (Seth Darst) によるThermus aquaticusのDNAポリメラーゼ結晶のX線回折像に基づいている。2008年現在、真正細菌のモデル生物である大腸菌のDNAポリメラーゼのX線結晶構造解析には成功していない。しかしながら、二次元結晶の電子顕微鏡で観察した大腸菌コアポリメラーゼの全体の形状は酷似しているため、詳細な構造も似ていると考えられている[20]。
真正細菌のコア酵素
T. Aquaticus のRNAポリメラーゼコア酵素はカニのはさみのようである。主にツメの一つはβサブユニット、もう一つはβ'サブユニットが占める。α1と2はヒンジにあり、それぞれβ、β'に結合している。小さなωサブユニットはβ'サブユニットのC末端[注釈 2]に巻きついており、はさみでいう峰に存在する。触媒活性中心 (catalytic center, 活性部位) は、βとβ'サブユニットの内部である活性中心溝 (channel) における付け根にある[21]。広さ約25 Åの空間である[22][23]。ここにはマグネシウムイオン (Mg2+) がβサブユニット中の3つのアスパラギン酸にキレート結合している。この3つはアミノ酸配列 NADFDGD(Dがアスパラギン酸)に含まれており、全ての細菌で保存されている[20]。
真正細菌のホロ酵素
2002年のダーストらのX線結晶構造解析から3つの結論が出された[24]。(1) σ因子(σA)とβおよびβ’サブユニットとの間には広い範囲の相互作用がある。(2) σ因子のN末端[注釈 2]にある91個のアミノ酸 (ドメイン1.1) が欠損しているホロ酵素にはDNAを通す割れ目があったが、それにしては小さい。このことから、91個のアミノ酸は割れ目をこじ開けてDNAを結合させると推測されている。(3) σ因子中のドメインのうちの2つ (ドメイン3と4) をつなぐ、明確な三次構造のないループはRNAポリメラーゼホロ酵素の活性部位に近く、また転写産物の出口に存在している。
2番目で欠損している部位を解釈しているのは、ダーストらは完全なホロ酵素を結晶化することができず、ドメイン1.1を欠損したσのそれを撮影に用いたからである[25]。よって、完全な構造は明らかでないが、その予測はできる。例えば、回折像によると切断されたN末端がαサブユニットの端に位置し、活性部位にまっすぐ向く。また、ドメイン1.1は中性pHで約3分の1の残基が負電荷となるほど酸性アミノ酸が非常に多い。塩基性アミノ酸が並ぶ活性部位にいかにも強く結合できそうである。ダーストらはこれを、ドメイン1.1は小さすぎる入口をこじ開けてDNAを内部に結合させるためと考えた[25]。そして、内部でDNAは融解し、ホロ酵素は閉鎖型複合体[注釈 3]になるのと考えられる。その際にドメイン1.1は解離し、内部のDNA周辺で活性部位は閉じると考えられる。この解離は、閉鎖型複合体に保護されていたのが、開放型複合体への移行でドメイン1.1がヒドロキシルラジカルにさらされるためのようである。リチャード・エブライトは閉鎖型複合体のドメイン1.1が開放型複合体では消えていることを蛍光共鳴エネルギー移動実験で証明した[25]。
3番目の見解には2つの解釈がある。第一に、σ因子は活性部位に近づくことでリン酸ジエステル結合の形成に携わる。第二に、ループの連結鎖は転写産物の出口を塞ぐことで、アボーティブ転写産物の形成を行う[注釈 3]。アボーティブ転写産物形成については、連結鎖と開始段階で合成されるRNAは出口を占有するための競合をするという仮説がある[26]。連結鎖が勝つとRNAの伸長は中断され、短いアボーティブ転写産物として放出される。アボーティブ転写産物は完成した転写産物より過剰に合成される (大腸菌では11倍過剰[26]) ので、この過程はおそらく何度も繰り返される。約12nt以上にうまく成長できたときにRNAはようやく競合に勝つ。連結鎖はRNAにどかされ、結果、コア酵素とσ因子との結合は弱くなる。もしくはコア酵素から解離して伸長への移行に備える。ダーストらは、連結鎖を欠損したσ因子でアボーティブ転写産物は多量に生産されないことを確認した[26]。アボーティブ転写産物はσ因子が活性部位に存在するための副産物であると推測される。伸長の礎となる短いDNAを結合させるためσ因子が活性部位に接近することで、必然的に連結鎖は出口を塞いでいると考えられる[26]。
真正細菌のホロ酵素-DNA複合体

ホロ酵素とDNAによって形成される複合体は、転写時の状態であるためRF複合体 (replicative form complex、RFは複製型) と呼ばれる。ダーストらは下図のフォークジャンクションDNAにT. aquaticus のDNAポリメラーゼホロ酵素を結合させた、RF複合体を作成した。このDNAは、-35ボックスを含むほとんどが二本鎖だが、-10ボックス中の非鋳型鎖[注釈 5]に-11位から始まる一本鎖の突出部分を持つ。これは開放型複合体における状態を模倣したものである (詳しくは#真正細菌のβサブユニット)。
RF複合体の立体構造から、様々な事実が判明した。ホロ酵素に結合するDNAはσサブユニットがある場所を横切る。大腸菌のプロモーターにおいては、-12位の塩基がσ70因子の領域2.4のGln437およびThr440と相互作用している。T. aquaticus のσAで2つのアミノ酸はGln260とAsn263とに相当する。
Trp256は-10ボックス直前の-12位に非常に近い。T. aquaticus σAのPhe248、Tyr253、Trp256や大腸菌σ70における一部の3 芳香族アミノ酸は高度に保存されている。これらは開放型複合体の-10ボックスの非鋳型鎖に結合することで、プロモーターの融解に関与すると予測される。観察されたTrp256の位置から-11位の塩基対の代わりとなり、融解を促進する可能性が高い。
σの領域2.2と2.3における2つの保存された塩基性アミノ酸(Arg237とLys241)が 静電相互作用で結合していることが観察された。しかし、領域4.2の残基は35ボックスに結合していない。ダーストらは、RF複合体の結晶化の際に、-35ボックスが領域4.2に対する正常な位置から押し出されてしまったと結論付けた[27]。ダーストらは自身の撮影したRF複合体の構造やその他の証拠から以下の仮説を提唱した[27]。DNAの上流で二本鎖DNAが曲がることによって、DNaseⅠの標的部位が生じる。一方、下流領域では二重らせんが融解する。こうして閉鎖型から開放型へと複合体が移行する。開放型複合体でのDNAや各タンパク質の相互作用も立体的に解析された。-10ボックスがβとβ‘サブユニットの間で融解するが、これはβ’舵型構造によって維持される。この構造はβ’サブユニットの表面から隣接するβサブユニットに向けて、また分離した2つのDNA鎖の間隙に突き出す。これによって、DNAの再会合は阻止される。
活性部位には2つのMg+が3つのアスパラギン酸によって支えられる。
非鋳型鎖 -40 -30 -20 -105' GGCCGC|TTGACA|AAAGTGTTAAATTG|TG|C|TATACT 3'3' CCGGCG|AACTGT|TTTCACAATTTAAC|AC|G|A 5' -35ボックス ↑ -10ボックス 拡張した-10ボックス 鋳型鎖図:RF複合体の作成に使用したDNA
少なくとも開放型複合体になった時点で、ホロ酵素には内部に通じる5つの通路がある[21]。NTP取り込み通路は基質であるリボヌクレオチドを触媒活性中心に迎え入れる。RNA出口通路は後の伸長段階で合成したRNA鎖の部分を出すためにある。ほかの3つの通路はDNAが出入りするために使う。下流のDNAは下流DNA用通路から二重らせんのまま活性中心溝に入る。そこでDNAは+3から2本の一本鎖に分かれる[28]。非鋳型鎖は非鋳型鎖用通路(NT通路)を抜けてホロ酵素の表面に沿って進む。一方、鋳型鎖は触媒活性溝を突き進み、鋳型鎖用通路(T通路)から外に出る。2つの一本鎖はホロ酵素の後方にある上流DNAの-11の位置で二重らせんに戻る[28]。
真核生物のRNAポリメラーゼ

真核生物にはRNAポリメラーゼI、II、IIIといった3種類のRNAポリメラーゼがある。1969年にロバート・ローダー (Robert Roeder) とウィリアム・ラター William Rutter が発見した[29]。3つは合成するRNAが異なり、RNAポリメラーゼⅠはrRNA前駆体を合成する。RNAポリメラーゼIIは、タンパク質をコードするmRNAのほか、いまだ謎の多いヘテロ核内RNA (heterogeneous nuclear RNA, hnRNA) や大部分の核内低分子RNA (small nuclear RNA, snRNA) を合成する。hnRNAとsnRNAは成熟mRNAの合成に関わる。RNAポリメラーゼIIIはtRNAや5S rRNA、前述とは別のいくつかのsnRNAの前駆体を担う。また、細胞内の分布も別で、RNAポリメラーゼIは核小体にだけ、IIとIIIが核質にだけ存在する。
細菌は開始因子が1つ (σ因子) だけだったが、真核生物では複数の基本転写因子 (general transcription factor, GTF) を必要とする。しかし、実際にはヌクレオソームがあるためさらにDNA結合調節タンパク、いわゆる介在複合体、ヌクレオソーム修飾酵素をはじめとしたいくつかのタンパク質を必要とする[30]。
RNAポリメラーゼIIのサブユニット
RNAポリメラーゼIIのサブユニット構成は、1971年にピエール・シャンボン (Pierre Chambon) らとラターらのグループから独立に報告された[31]。この時は不完全だったが、1975年にマウス由来の全てのRNAポリメラーゼから、ローダーらがほぼ完全な情報を明らかにした[31]。現在では全3種のサブユニットについて正確に判明している。
ヒトと酵母におけるポリメラーゼIIの12個のサブユニットについて下の表にまとめた。これらは各々単独の遺伝子にコードされている。各サブユニットの名前はその遺伝子の名前に由来する。RPBという名称は、シャンボンが用いたRNAポリメラーゼB (すなわちII) という呼び名にちなむ。
リチャード・ヤング (Richard Young) はエピトープタグ法で同定した10個のサブユニットを3つに分類した[31]。真正細菌のRNAポリメラーゼコア酵素に構造・機能ともに類似するコアサブユニット、少なくとも酵母では3種類の核内RNAポリメラーゼ全てにある共通サブユニット (common subunits)、必ずしも酵素活性にいつも必要ではない非必須サブユニットの3つである。
電気泳動の結果から、Rpb1サブユニットには215 kDのIIaと240 kDと測定されたIIoの2つの形態が存在する。IIaのC末端にはCTD (carboxyl-terminal domain) と呼ばれる、7個のアミノ酸(heptad)から成る共通配列 Tyr-Ser-Pro-Thr-Ser-Pro-Ser が反復した配列がある。IIoはCTDのヒドロキシ基を持ったアミノ酸(セリン、スレオニン、チロシン)がリン酸化したものである。しかし、哺乳類のhaptadは52回反復するが、これが全てリン酸化したとしてもIIaとIIoの分子量差を埋めることはできない。実際の分子量が大きく見えるよう、泳動度が遅くなるよう、リン酸化は立体構造の変化を引き起こすと考えられている。異なるRpb1サブユニットを所有するRNAポリメラーゼIIをそれぞれRNAポリメラーゼIIA (RNA polymerase IIA) およびRNAポリメラーゼIIO (RNA polymerase IIO) と区別する。前者はプロモーターに最初に結合するときの形態で、後者は伸長反応を行う。
| サブユニット | 酵母遺伝子 | 酵母タンパク質のモル質量(kD) | 特徴 |
|---|---|---|---|
| hRPB1 | RPb1 | 192 | コアサブユニット。CTDを含み、DNAと結合する。プロモーターの選別に関与。β’と相同。 |
| hRPB2 | RPb2 | 139 | 活性部位を含むコアサブユニット。プロモーターの認識と伸長速度に関与。β’に相同。 |
| hRPB3 | RPb3 | 35 | コアサブユニット。原核生物のαサブユニットと相同で、Rpb11と機能する可能性あり。 |
| hRPB4 | RPb4 | 25 | 非必須サブユニット。Rpb7と複合体を形成し、ストレス応答に関与する。 |
| hRPB5 | RPb5 | 25 | 共通サブユニット。転写アクチベーターの標的。 |
| hRPB6 | RPb6 | 18 | 共通サブユニット。複合体形成と安定化に寄与。 |
| hRPB7 | RPb7 | 19 | 定常期のRpb4と複合体を形成。 |
| hRPB8 | RPb8 | 17 | 共通サブユニット。オリゴヌクレオチド/オリゴ糖結合ドメイン。 |
| hRPB9 | RPb9 | 14 | 伸長に関与する可能性があるZnリボンモチーフを含む。プロモーターを認識。 |
| hRPB10 | RPb10 | 8 | 共通サブユニット。 |
| hRPB11 | RPb11 | 14 | 原核生物のαサブユニットと相同で、Rpb3と機能する可能性あり。 |
| hRPB12 | RPb12 | 8 | 共通サブユニット。 |
RNAポリメラーゼIIの立体構造
ロジャー・コーンバーグらは2001年に X線構造解析の結果を発表した[33]。RNAポリメラーゼIIの結晶化は難しく、撮影に用いたのはRpb4とRpb7を欠いた酵母変異株のもの(polII Δ4/7)だった。これは転写を開始できないが、伸長反応は問題なくできる。
全体の構造は巨大な顎のようで、酸性のDNAをくわえる深い溝がある。このため残りの酵素表面は酸性であるのに対し、溝には塩基性残基が並ぶ。上顎はRpb1とRpb9、下顎はRpb5である。底の触媒活性中心には2個のMg2+があり、コーンバーグらはメタルA (metal A) とメタルB (metal B) に区別した[34]。メタルAはRpb1のD481、D483、D485といった3個のアスパラギン酸と強固に結合している。一方、メタルBはRpb1のD481、Rpb2のE836とD837に囲まれているものの、配位結合するには距離がある。触媒反応の過程でこれら酸性アミノ酸が近づくと考えられる。メタルBは基質のリボヌクレオチド三リン酸と結合する。
真正細菌同様、RNAポリメラーゼIIにもポア1 (pore 1) という、合成したRNAを出す出口が存在する。漏斗状のポア1外縁には、出てきたRNAを切断するTFIISと結合するアミノ酸が並ぶ[34]。一方、入り口は14 Åにも及ぶクランプモジュール (clamp module) が回転することによって開閉される[35]。プロモーターは酵素表面でほどかれ、相補鎖を外に残して鋳型鎖が溝の中へ誘導される。
RNAポリメラーゼIIの伸長複合体
コーンバーグらはDNAと合成したRNA両方と結合したRNAポリメラーゼIIの撮影にも成功した[35]。単独でクランプモジュールは開いて外から活性中心に近づけたが、伸長複合体のクランプモジュールは閉じ、鋳型鎖と転写産物を覆う[36]。後述するように、転写中のDNAは内部で折れ曲がらなければならない。しかし、転写が開始する前のDNAは比較的強固なまっすぐな構造をしている[37]。最初にDNAを入れるときは開いているが、途中からDNAが酵素から離れないように閉じるのである。メタルAは、最近付加された2つのリボヌクレオチド間のリン酸に結合できる位置にある[36]。活性中心の近くには溝にまたがったブリッジヘリックス (bridge helix) が観察される。まっすぐに伸びた状態では基質のリボヌクレオチド三リン酸が入れるようポア1は開いている。一方で、Thr831とAla832の付近で曲がる状態もあり、活性中心は閉ざされる[36]。
内部のDNAは入口の所でその先にある壁のために無理やり曲げられる。酵素表面でほどかれた鋳型鎖はRNAと二重らせん形成するが、この長さはラダー (rudder, 舵) と呼ばれるタンパク質が障害物となり9bpに制限される[37]。それ以上付加されると、塩基対形成している最後のリボヌクレオチドがDNAから離れ、RNAの出口から抜け出す。DNAも別の出口で脱出し、鋳型鎖と非鋳型鎖は二重らせんに戻る。RNAポリメラーゼの進路、DNAの下流を前とするなら、後ろの壁から上にRNA・DNA出口が、下にポア1が開いている[37]。
注釈
出典
関連項目
- 転写 (生物学)
- DNAポリメラーゼ - DNAやRNAを鋳型として相補的なDNA鎖を合成する酵素の総称
- DNAプライマーゼ - DNAを鋳型として相補的なRNA断片(プライマー)を合成する酵素の総称
- RNA依存性RNAポリメラーゼ - RNAを鋳型として相補的なRNA鎖を合成する酵素
