SARS-CoV-2
- Denne artikel er om et virus. For sygdommen se COVID-19. For udbruddet af denne virus, se coronaviruspandemien i 2019-2020.
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2),[1][2] tidligere midlertidigt navngivet 2019-novel-coronavirus eller 2019-nCoV[3][4] og uformelt kendt som Wuhan-coronavirus,[5][6]er en ny type Corona-virus i familie med SARS. SARS-CoV-2 er en smitsom virus, som kan forårsage COVID-19, en luftvejsinfektion, som kan udvikle sig til Langvarig COVID-19.
| Novel Coronavirus (SARS-CoV-2) | |
|---|---|
 Novel Coronavirus (SARS-CoV-2). | |
| Videnskabelig klassifikation | |
| Domæne | Vira (Virus) |
| (urangeret) | Gruppe IV (+ssRNA) |
| Orden | Nidovirales |
| Familie | Coronaviridae |
| Slægt | Coronavirus |
| Arter | |
| |
| Hjælp til læsning af taksobokse | |
SARS-CoV-2 blev oprindeligt identificeret i midten af december 2019 i millionbyen Wuhan i det centrale Kina efter mange mennesker fik lungebetændelse af ukendte årsager, primært knyttet til dem, der arbejdede på Huanan Seafood Market (華南 海鮮 市場), hvor der sælges levende dyr. Kinesiske forskere isolerede derefter den nye coronavirus SARS-CoV-2, som har vist sig at være mindst 70 % identisk med gensekvensen hos SARS-CoV. Den har dog endnu ikke vist sig at være så alvorligt eller så dødbringende som SARS.20. januar 2020 bekræftede de kinesiske myndigheder og WHO, at virussen kan smitte fra menneske-til-menneske,[7] Og med 7.700 personer smittet og 170 dødsfald erklærede WHO d. 30. januar 2020 den potentielt dødelige SARS-CoV-2 for en international sundhedskrise.[8]I slutningen af januar vurderede kinesiske myndigheder virusset til at være en kategori B-sygdom på linje med AIDS, SARS og polio. Ikke desto mindre håndterede myndighederne det som en kategori A-sygdom, som Kina ellers kun har to af, nemlig kolera og byldepest.[9]
Oprindelse
Den 22. januar 2020 udgav Journal of Medical Virology en rapport med en genomisk analyse, som overvejer at slanger i Wuhan-området er citat: "the most probable wildlife animal reservoir" for virussen. Mere forskning på området er dog påkrævet.[10][11]
En homolog rekombinationshændelse kan have blandet en "clade A"-virus (flagermus SARS-lignende vira CoVZC45 og CoVZXC21) med RNA-binding af en indtil videre ukendt Beta-CoV.[12][13]
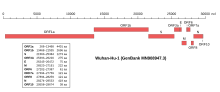
Isolaten Wuhan-Hu-1 (GenBank-nummer MN908947[14]) af SARS-CoV-2 viser store fylogenetiske ligheder med to coronavirus-isolater fra kinesiske flagermus, som blev karakteriseret i 2015 og 2017.[15]
Isolatens virusgenom omfatter 29.875 bp med 281 bp henholdsvis 325 bp lange uoversatte områder ved 5'-enden og henholdsvis 3'-enden. De formodede kodende områder fordeler sig på 10 proteiner: et 7096 aminosyre langt ORF1ab-polyprotein, et 1282 aminosyre langt overflade-glykoprotein kaldet spike, et 75 aminosyre hylsterprotein (E), et 222 AS membranglykoprotein (M), et 419 AS nukleokapsid-fosfoprotein og yderligere 5 proteiner (ORF3a, ORF6, ORF7a, ORF8 og ORF10). Disse udgør genfølgen i SARS-virus og alle coronavira.[16]
Varianter
Helgenomssekventering af SARS-CoV-2 og indføring i databaser kan give et overblik over genetiske mutation i virussens genom.GISAID-databasen indeholder sådanne genomdata og analyser af genomet ses også i forbindelse med Nextstrain.[17]
Det findes [mindst] titusindvis[18] af varianter af virusset.[19] I Norden har man fundet varianter af virusset; således har man i Norge konstateret "over 40"[20] variationer.[21]
En person med svækket immunsystem fik igennem sit COVID-19-sygdomsforløb en længere række af mutationer, særligt i spike-proteinet.[22]
Før Omicron-varianten viste sig i mennesker i Sydafrika i 2021, kan den have udviklet sig i dyr. Det usædvanligt store antal mutationer sammenlignet med den originale variant - omkring 50, inklusive mere end 30 i Spike-proteinet, dvs. næsten tre gange så mange som Delta-varianten - antyder en udvikling i en usædvanlig vært, jf. minksagen og zoonose.
- Notable varianter
- Cluster 5: I Danmark er der indtil 5. november 2020 fundet fem typer af varianter af SARS-CoV-2 i mink. Statens Serum Institut (SSI) har navngivet dem som cluster 1-5. Blandt disse varianter er der konstateret syv forskellige mutationer i virussets såkaldte spike-protein på virussets overflade. Der har været ændringer i aminosyrerne H69del/V70del, Y453F, I692V og M1229I.[23][24][25][26][27]
- B.1.1.207[28][29] er en variant som har en P681H mutation, som også findes i VOC-202012/01 fra Storbritannien.[29]
- B.1.1.7[30] også kendt som VOC 202012/01[31][32][33][34] eller 20B/501Y.V1.[35][36][37][38][39] Variant fra denne udviklingslinje blev først identificeret i prøve fra den sydvestlige del af England.[40] Varianten blev [først] kaldet VUI – 202012/01[41].
- B.1.1.317[42]
- B.1.617 er den dominerende variant i Indien [63]
- B.1.617.2. varianten kendes som “delta” og er mere smitsom [64]
- udviklingslinje P.1 blev opdaget i Tokyo d. 6. januar 2021 af Kokuritsukansenshōkenkyūjo (NIID). Variant fra denne udviklingslinje blev først identificeret i fire personer, der ankom Tokyo efter at have rejst fra Amazonas (delstat i Brasilien), d. 2. januar 2021.[65][66]
| First detection | Klassificering (Rambaut et al.) | Andre navne | Notable mutationer | Ref. | |||||
|---|---|---|---|---|---|---|---|---|---|
| Sted | Dato | ||||||||
 Nigeria Nigeria | aug 2020 | B.1.1.207 | P681H | [2] | |||||
 Storbritannien Storbritannien | sep 2020 | B.1.1.7 | VOC-202012/01, 20I/501Y.V1 | N501Y, 69–70del, P681H | [2][67][68][69] | ||||
 Danmark Danmark | okt 2020 | B1.1.298 | Cluster 5, ΔFVI-spike (SSI), Covid-19-minkvarianter, DCGC-3024/2020, en af fem danske mink-varianter (cluster1-5) | Y453F, I692V, M12291I, 69–70deltaHV | [24][26][27] | ||||
 Sydafrika Sydafrika | dec 2020 | B.1.351 | 501.V2, 20H/501Y.V2, VOC-202012/02 | N501Y, K417N, E484K | [2][50][70][67][71][72][73] | ||||
 Storbritannien Storbritannien Nigeria Nigeria | dec 2020 | B.1.525 | VUI-202102/03 (PHE), tidligere UK1188 | E484K, F888L | [70][62][59] | ||||
 USA USA | jan 2021 | B.1.429 | Q677H, S:Q677H, 677, Q677, B.1.429+S:Q677H[74] | [75][76][77] | |||||
 Japan Japan Brasilien Brasilien | jan 2021 | P.1 | Stammer fra B.1.1.28 | N501Y, E484K, K417T | [78][79][65][80][67] | ||||
 USA USA | mar 2021 | Q677P | 677 eller Q677P stammer fra B.1.429[81][82] | “Pelican”, “Robin” (Robin 1, Robin 2), “Yellowhammer”, ”Bluebird”, ”Quail” og “Mockingbird”[83] | [84][76][85] | ||||
 Storbritannien Storbritannien | apr 2021 | B.1.1.7 | B1.1.7, “Cluster B117” | [86][62][87] | |||||
 USA USA | mar 2021 apr 2021 | B1.526 | 526 | B1 (526-537) (STJ72853), P.1; P.2. | [88][76][89] | ||||
I nyhedsmedierne blev flere varianter navngivet efter det land, som varianten først blev konstateret i, for eksempel den "britiske variant" eller "engelske variant" for (B.1.1.7) og senere blandt andre også en "sydafrikansk" (B.1.351) og en "indisk" (B.1.617.2). Den Indiske regering beklagede i maj 2021, at mutationer fik landenavne. Den 31. maj besluttede WHO at mutationerne i stedet skulle benævnes med bogstaver fra det græske alfabet, således kom den "britiske variant" til af hedde "alpha", den "sydafrikanske variant" blev til "beta" og den "indiske variant" benævnt som "delta".[90]
Notable missense-mutationer
- D614G: En af de betydelige ændringer i SARS-CoV-2-genomet under COVID-19-pandemien forekom i receptorbindingsdomænet, nærmere bestemt spike-proteinets D614G-mutation. D614-varianten dominerede oprindeligt men blev udkonkurreret af G614-varianten.[91]
Nomenklatur af varianter
| Pango udviklingslinjer | Rambaut et al.[106] | Nextstrain klade, 2021[107] | GISAID klade | Notable varianter |
|---|---|---|---|---|
| A.1–A.6 | 19B | S | inkluderer "sekvens null" (sequence zero)[108] | |
| B.3–B.7, B.9, B.10, B.13–B.16 | 19A | L | ||
| O[a] | ||||
| B.2 | V | |||
| B.1 | B.1.5–B.1.72 | 20A | G | Udviklingslinje B.1 hos Rambaut et al. |
| B.1.9, B.1.13, B.1.22, B.1.26, B.1.37 | GH | |||
| B.1.3–B.1.66 | 20C | Inkluderer CAL.20C[55] | ||
| 20G | ||||
| 20H | I 501.V2 også kendt som | |||
| B.1.1 | 20B | GR | Inkluderer B.1.1.207 | |
| 20D | Inkluderer P.1 og P.2[109] | |||
| 20F | ||||
| 20I | Inkluderer VOC-202012/01 også kendt som | |||
| B.1.177 | 20E (EU1)[107] | GV[a] | Fra 20A[107] |
Se også
Kilder/henvisninger
Eksterne henvisninger
Fodnotefejl: <ref>-tags eksisterer for en gruppe betegnet "lower-alpha", men der blev ikke fundet et tilsvarende {{reflist|group="lower-alpha"}}, eller et afsluttende </ref>-tag mangler
