CRISPR (edición de xenes)
Ferramentas
Xeral
Imprimir/exportar
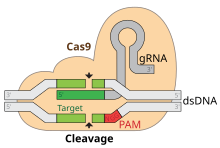
A edición de xenes por CRISPR (adoita pronunciarse crísper) é unha técnica de enxeñaría xenética en bioloxía molecular por medio da cal pode modificarse o xenoma dos organismos vivos. CRISPR é a abreviatura en inglés de Clustered Regularly Interspaced Short Palindromic Repeats, repeticións palindrómicas curtas agrupadas e espazadas regularmente, unha familia de secuencias de ADN dos procariotas derivadas de fragmentos de bacteriófagos que as infectaran. A técnica está baseada nunha versión simplificada do sistema de defensa antiviral bacteriano CRISPR-Cas9, usado polos procariotas para destruír ADN de bacteriófagos. Con este método, ao introducir nunha célula a nuclease Cas9 en complexo cun ARN guía (ARNg ou, en inglés, gRNA) sintético, pode cortarse o xenoma da célula no lugar desexado, o que permite eliminar xenes existentes ou engadir xenes novos in vivo.[1]
Esta técnica considérase moi importante en biotecnoloxía e medicina, xa que permite editar xenomas in vivo con gran precisión, facilidade e baixo custo. Pode utilizarse na creación de novas medicinas, produtos agrícolas e organismos modificados xeneticamente, ou como medio de controlar patóxenos e pragas. Tamén ten posibilidades no tratamento de enfermidades conxénitas herdadas xeneticamente, así como de doenzas que se orixinan por mutacións somáticas, como o cancro. Porén, o seu uso na modificación xenética da liña xerminal humana é moi polémica. O desenvolvemento da técnica serviulles a Jennifer Doudna e Emmanuelle Charpentier para gañar o Premio Nobel de Química de 2020.[2][3] O terceiro grupo de investigadores que compartiu o premio Kavli polo mesmo descubrimento,[4] liderado por Virginijus Šikšnys, non foi galardoado co premio Nobel.[5][6][7]
A nuclease Cas9, que funciona como tesoira xenética, abre ambas as febras dunha secuencia diana de ADN para introducir a modificación por medio dun de dous posibles métodos. As mutacións knock-in, facilitadas por reparación dirixida por homoloxía (HDR), son a vía tradicional usada polas estratexias de edición xenómica dirixida a unha diana.[1] Isto permite a introdución de danos e reparacións no ADN dirixidas a unha diana. A HDR emprega secuencias de ADN similares para reparar as roturas por incorporación de ADN exóxeno que funciona como molde para a reparación.[1] Este método depende de que ocorran danos no ADN periódicos e illados no sitio diana para que comece a reparación. As mutacións knock-out causadas por CRISPR-Cas9 orixínanse pola reparación da rotura de dobre febra no ADN por medio de unión de extremos non homólogos (NHEJ) ou unión de extremos mediada pola POLQ/ polimerase theta (TMEJ). Estas dúas vías de unión de extremos poden con frecuencia orixinar delecións ou insercións aleatorias no sitio de reparación, o cal pode impedir ou alterar o funcionamento do xene. Polo tanto, a enxeñaría xenómica por CRISPR-Cas9 dálle aos investigadores a capacidade de xerar a alteración aleatoria dun xene diana. Debido a isto, a precisión da edición do xenoma é unha gran preocupación. A edición do xenoma orixina cambios irreversibles no xenoma.
Aínda que a edición xenómica en células eucariotas foi posible usando varios métodos desde a década de 1980, os métodos empregados foran ineficientes e pouco prácticos para aplicalos a grande escala. Co descubrimento de CRISPR e especificamente a molécula de nuclease Cas9, a posibilidade dunha edición eficiente e moi selectiva é agora unha realidade. A Cas9 derivada da especie bacteriana Streptococcus pyogenes facilitou a modificación xenómica dirixida a diana en células eucariotas ao permitir un método fiable de crear unha rotura dirixida a un lugar específico designado polas febras guía de ARNtracr (en inglés tracrRNA) e o ARNcr (en inglés crRNA).[8] A facilidade coa cal os investigadores poden inserir a Cas9 e o molde de ARN para silenciar ou causar mutacións puntuais en loci específicos é moi valiosa para un rápido e eficiente mapado de modelos xenómicos e procesos biolóxicos asociados con varios xenes en diversos eucariotas. Desenvolvéronse variantes novas feitas por enxeñaría da nuclease Cas9 que reducen significativamente a actividade fóra de diana.[9]
As técnicas de edición do xenoma CRISPR-Cas9 teñen moitas aplicacións potenciais, incluíndo en medicina e agricultura. O uso do complexo CRISPR-Cas9-ARNg para a edición de xenomas[10] foi elixido pola Asociación Americana para o Avance da Ciencia (AAAS) como descubrimento do ano en 2015.[11] Expresáronse moitas cuestións bioéticas sobre a perspectiva do uso de CRISPR para a edición da liña xerminal, especialmente en embrións humanos.[12]
A inicios da década de 2000 investigadores alemáns empezaron a desenvolver as nucleases de dedo de cinc (ZFNs), que son proteínas sintéticas cuxos dominios de unión ao ADN lles permiten crear roturas de dobre febra no ADN en sitios específicos. As nucleases de dedo de cinc teñen unha maior precisión e a vantaxe de ser menores que Cas9, pero non se usan tan comunmente coma os métodos baseados en CRISPR. En 2010, as nucleases sintéticas chamadas nucleases efectoras similares a un activador da transcrición (TALENs) proporcionaron un modo máis doado de tomar como diana unha rotura de dobre febra nunha localización específica da febra de ADN. Tanto as nucleases de dedo de cinc coma as TALENs requiren o deseño e creación dunha proteína á medida para cada secuencia diana de ADN, o cal é un proceso moito máis difícil e leva máis tempo que deseñar ARNs guías. Os CRISPRs son moito máis fáciles de deseñar porque o proceso require sintetizar só unha curta secuencia de ARN, un procedemento que xa se usa moito noutras técnicas de bioloxía molecular (por exemplo, para crear cebadores oligonucleótidos).[13]
Mentres que métodos como a interferencia de ARN (RNAi) non suprimen totalmente a función xénica, CRISPR, as ZFNs e as TALENs proporcionan un knockout de xenes completo e irreversible.[14] CRISPR pode tamén tomar como diana varios sitios do ADN simultaneamente simplemente introducindo diferentes ARNg. Ademais, o custo de empregar CRISPR é relativamente baixo.[14][15][16]
En 2012 Jennifer Doudna, Emmanuelle Charpentier, Martin Jínek, Krzysztof Chyliński, Ines Fonfara e Michael Hauer publicaron os seus descubrimentos de que o CRISPR-Cas9 podía programarse con ARN para editar o ADN xenómico, agora considerado un dos descubrimentos máis significativos na historia da bioloxía.[17][18]
As patentes parqa CRISPR pasaron por varias vicisitudes.
En novembro de 2013, SAGE Labs (parte do grupo Horizon Discovery) tiña dereitos exclusivos dunha desas compañías para producir e vender ratos modificados xeneticamente e dereitos non exclusivos sobre modelos de ratos e coelllos.[19] En 2015 Thermo Fisher Scientific tiña licenza de propiedade intelectual de ToolGen para desenvolver kits de reactivos CRISPR.[20]
En decembro de 2014 os dereitos de patente para CRISPR foron impugnados. Formáronse varias compañías para desenvolver fármacos e ferramentas de investigación.[21] A medida que as compañías incrementaron o seu financiamento, empezaron a formularse dúbidas sobre se o CRISPR podería ter un rendemento económico rápido.[22] En 2014 Feng Zhang do Broad Institute do MIT e Harvard e outros nove recibiron a patente estadounidense número 8.697.359[23] sobre o uso da edición de xenes por CRISPR–Cas9 en eucariotas. Aínda que Charpentier e Doudna (grupo CVC) foron recoñecidos como os que concibiron CRISPR, o Broad Institute foi o primeiro que conseguiu unha "redución á práctica" de acordo cos xuíces e xuízas da patente Sally Gardner Lane, James T. Moore e Deborah Katz.[24]
O primeiro conxunto de patentes concedéuselle ao equipo do Broad Institute en 2015, o que motivou que os avogados do grupo CVC interpuxesen o primeiro procedemento de interferencia.[25] En febreiro de 2017 a Oficina de Patentes dos EUA (USPTO) xulgou un caso de interferencia de patente presentado pola Universidade de California con respecto a patentes concedidas ao Broad Institute, e atopou que as patentes de Broad, que afirmaban comprendian a aplicación de CRISPR-Cas9 en células eucariotas, eran distintas das invencións aducidas pola Universidade de California.[26][27][28]
Pouco despois, a Universidade de California presentou unha apelación a este pronunciamento.[29][30] En 2019 iniciouse un segundo preito de interferencia. Foi unha resposta a aplicacións da patente feitas por CVC que solicitou ao tribunal de apelación determinar cal era o inventor orixinal desta tecnoloxía. A USPTO sentenciou en marzo de 2022 contra a Universidade de California, establecendo que o Broad Institute foi o primeiro en rexistrala. A decisión afectou moitos dos acordos de licenzas para a tecnoloxía de edición por CRISPR que foran concedidas pola Universidade de Californa, Berkeley. A Universidade de California declarou a súa intención de apelar o dictame ao USPTO.[31]
En marzo de 2017 a Oficina de Patentes Europea (OPE) anunciou a súa intención de dar dereito de editar todos os tipos de células ao Instituto Max-Planck de Berlín, á Universidade de California e á Universidade de Viena,[32][33] e en agosto de 2017, a OPE anunciou a súa intención de dar dereitos sobre CRISPR nunha aplicación de patente presentada por MilliporeSigma.[32] En agosto de 2017 a situación da patente en Europa era complexa, con MilliporeSigma, ToolGen, a Universidade de Vilnius e Harvard disputándose os dereitos, xunto coa Universidade de Califora e Broad.[34]
En xullo de 2018, o Tribunal Europeo de Xustiza sentenciou que a edición de xenes de plantas era unha subcategoría dos alimentos OMG e, polo tanto, que a técnica CRISPR sería regulada de aí en adiante na Unión Europea polas súas regras e regulacións para os organismos modificados xeneticamente (OMG).[35]
En febreiro de 2020, un ensaio en EUA mostrou que a edición de xenes por CRISPR era segura en tres pacientes de cancro.[36]
En outubro de 2020 as investigadoras Emmanuelle Charpentier e Jennifer Doudna recibiron o Premio Noble de Química polo seu traballo no campo dos CRISPR.[37][38] Fixeron historia por ser as dúas primeiras mulleres que compartiron este premio sen compartilo tamén cun home.[39][5]
En xuño de 2021 concluíu un primeiro pequeno ensaio clínico da edición de xenes por CRISPR intravenosa en humanos con resultados prometedores.[40][41]
En setembro de 2021 o primeiro alimento editado con CRISPR saíu á venda pública no Xapón. Eran uns tomates modificados xeneticamente para teren unhas cinco veces a cantidade normal do neurotransmisor con posible efecto calmante[42] GABA (un neurotransmisor inhibitorio).[43] CRISPR fora aplicado por primeira vez en tomates en 2014.[44]
En decembro de 2021 informouse de que o primeiro alimento animal mariño con edición de xenes por CRISPR e un un segundo conxunto de alimentos editados con CRISPR saíron á venda no Xapón: creáronse dous peixes, un dos cales crecía case o dobre do normal, pola alteración da leptina, a cal controla o apetito, e o outro crecía ao 1,2 do seu tamaño natural coa mesma cantidade de comida debido á alteración da miostatina, que inhibe o crecemento muscular.[45][46][47]
Un estudo de 2022 sobre a preferencia dos consumidores de comprar tomates CRISPR atopou que darlles a coñecer máis sobre os tomates CRISPR tivo un forte efecto positivo sobre a prefeencia dos participantes. "Case a metade dos 32 participantes de Alemaña que eran científicos demonstraron eleccións constantes, mentres que a maioría mostrou un aumento na súa intención de comprar tomates CRISPR, principalmente non científicos."[48][49]
A Universidade de California, Berkeley anunciou en maio de 2021 o seu intento de poxa de token non funxible de ambas as patentes de edición de xenes por CRISPR así como da inmunoterapia do cancro, aínda que a universidade conservaría neste caso a propiedade das patentes.[50][51] O 85 % dos fondos reunidos polas vendas da colección chamada The Fourth Pillar (O cuarto piar) ían ser utilizados para financiar investigacións.[52][53] Vendeuse en xuño de 2022 por 54.000 dólares.[54]

A edición do xenoma por CRISPR-Cas9 lévase a cabo cun sistema CRISPR de tipo II. Cando se utiliza para a edición do xenoma, este sistema inclúe unha ribonucleoproteína (RNP), que consta de Cas9, ARNcr (en inglés crRNA), e ARNtracr (en inglés tracrRNA), xunto cun molde para a reparación do ADN opcional.

| Compoñente | Función |
|---|---|
| ARNcr (ARN CRISPR) | Contén o ARN guía que localiza o segmento correcto do ADN do hóspede xunto cunha rexión que se une ao ARNtracr (xeralmente nunha forma de bucle en forquita), formando un complexo activo. |
| ARNtracr (ARNcr transactivante) | Únese ao ARNcr e forma un complexo activo. |
| ARNgs (sgRNA en inglés) | Os ARNs guías simples ou únicos (single guide RNA) son uns ARNs combinados que constan dun ARNtracr e polo menos un ARNcr. |
| Cas9 (o máis común) | Un encima cuxa forma activa pode modificar o ADN. Existen moitas variantes con diferentes funcións (é dicir, encima que forma unha amosega nunha soa das febras, que crea roturas de dobre febra, que se une ao ADN) debido á función do sitio de recoñecemento do ADN de cada encima. |
| Molde de reparación | Molécula de ADN usada como molde no proceso de reparación do ADN da célula hóspede, que permite a inserción dunha secuencia de ADN específica no segmento hóspede roto por Cas9. |
O CRISPR-Cas9 a miúdo emprega plásmidos, que codifican os compoñentes da RNP, para a transfección das células diana, ou a RNP ensámblase antes da adición ás células por medio de nucleofección.[55] Os principais compoñentes deste plásmido están representados na imaxe e listados na táboa. O ARNcr deséñase expresamente para cada aplicación, xa que esta é a secuencia que a Cas9 usa para identificar e unirse directamente a secuencias específicas dentro do ADN da célula hóspede. O ARNcr debe unirse soamente no lugar que se desexa. O molde de reparación é tamén deseñado expresamente para cada aplicación, xa que debe complementar ata certo punto as secuencias de ADN a cada lado do corte e tamén contén calquera secuencia que se desexe para a inserción no xenoma hóspede.
Poden empaquetarse xuntos múltiples ARNcr e o ARNtracr para formar un ARN guía simple ou único (ARNgs).[56] Este ARNgs pode incluírse xunto ao xene que codifica a proteína Cas9 e formar un plásmido para ser transfectado ás células. Disponse de moitas ferramentas on line para axudar no deseño de secuencias de ARNgs efectivas.[57][58]

Entre as proteínas alternativas a Cas9 están as seguintes:
| Proteína | Principal uso / características | Ano/s |
|---|---|---|
| Cas12 | Cas12a é máis pequena e simple que Cas9; Cas12b i.a. para a enxeñaría xenética en plantas[59][60] | |
| Cas13 | para a edición do ARN[61] | |
| Cas3[62][63] | Crea un amplo tramo monocatenario[64] | 2019 |
| CasMINI | Unhas dúas veces máis compacta que a máis frecuentemente usada Cas9 e Cas12a.[65][66] | 2021 |
| SuperFi-Cas9 | Máis exacta sen deceleración da velocidade[67][68] | 2022 |
| Cas7-11 | Máis axeitada para a edición do ARN terapéutica que a Cas13[69] | 2022 |
| Reparación do ADN tomando como molde o cromosoma homólogo | Este método só é aplicable a organismos cuxo cromosoma correspondente ten os xenes desexados.
| 2022 |
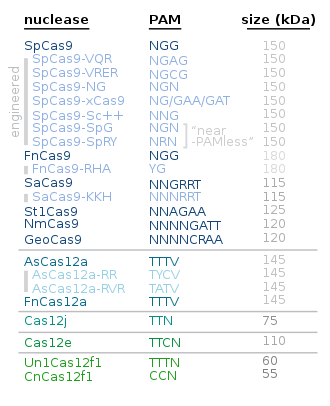
CRISPR-Cas9 ofrece un alto grao de fidelidade e a súa construción é relativamente simple. Depende de dous factores para a súa especificidade: a secuencia diana e a secuencia motivo adxacente protoespazador (PAM, do inglés protospacer adjacent motif). A secuencia diana é dunha lonxitude de 20 bases como parte de cada locus CRISPR na matriz de ARNcr.[55] Unha matriz de ARNcr típica ten múltiples secuencias diana únicas. As proteínas Cas9 seleccionan a localización correcta no xenoma do hóspede utilizando a secuencia para unirse cos pares de bases do ADN hóspede. A secuencia non forma parte da proteína Cas9 e como resultado é personalizable e pode ser sintetizada independentemente.[72][73]
A secuencia PAM no xenoma hóspede é recoñecida por Cas9. Cas9 non pode ser doadamente modificada para recoñecer unha secuencia PAM diferente. Porén, isto no fin de contas non é demasiado limitante, xa que é tipicamente unha secuencia moi curta e non específica que aparece frecuentemente en moitos lugares por todo o xenoma (por exemplo, a secuencia PAM SpCas9 é 5'-NGG-3', que no xenoma humano aparece aproximadamente cada 8 a 12 pares de bases).[55]
Unha vez que estas secuencias foron ensambladas nun plásmido e transfectadas nas células, a proteína Cas9 coa axuda do ARNcr encontra a secuencia correcta no ADN da célula hóspede e (dependendo da variante da Cas9) crea unha rotura do febra simple ou dobre na localización apropiada no ADN.[74]
As roturas dunha soa febra espazadas adecuadamente no ADN hóspede poden activar a reparación dirixida por homoloxía, a cal é menos tendente ao erro que a unión de extremos non homólogos que segue tipicamente a unha rotura de dore febra. Proporcionar un molde para a reparación permite a inserción dunha secuencia de ADN específica nunha localización exacta dentro do xenoma. O molde de reparaión debería estenderse de 40 a 90 pares de bases alén da rotura inducida por Cas9.[55] O obxectivo é que o proceso de reparación dirixida por homoloxía da célula utilice o molde de reparación proporcionado e, por tanto, incorpore a nova secuencia no xenoma. Unha vez incorporada, esta nova secuencia é agora parte do material xenético da célula e transmítese ás células fillas. A inhibición transitoria combinada da unión de extremos non homólogos por unha molécula de ARN interferente pequeno pode incrementar a eficiencia da reparación dirixida por homoloxía ata nun 93 % e impedir simultaneamente a edición fóra de diana.[75]
A entrega ás células da Cas9, o ARNgs, e os complexos asociados pode realizarse por medio dun virus ou por sistemas non virais. A electroporación do ADN, ARN ou ribonucleocomplexos é unha técnica común, aínda que pode ter efectos daniños nas células diana.[76] Utilizáronse técnicas de transfección química que utilizan lípidos e péptidos para introducir nas células os ARNgs en complexo con Cas9.[77][78] A entrega baseada en nanopartículas tamén se utilizou para a transfección.[79] Os tipos de células que son máis difíciles de transfectar (por exemplo, células nais, neuronas e células hematopoéticas) requiren sistemas de entrega máis eficientes, como os baseados en lentivirus (LVs), adenovirus (AdV) e virus adeno-asociados (AAV).[80][81][82]
A eficiencia da CRISPR-Cas9 increméntase moito cando varios compoñentes do sistema incluíndo a estrutura CRISPR/Cas9 enteira e os complexos Cas9-ARNg son entregados ensamblados en vez de usando métodos transxénicos.[83][84] Isto ten especial valor en plantas agrícolas modificadas xeneticamente para a comercialización en masa.[85][86] Como non se necesita a maquinaria de replicación do hóspede para producir estas proteínas, as posibilidades da secuencia de recoñecemento do ARNgs son case nulas, diminuíndo a probabilidade de efectos fóra de diana.[79]
Outras melloras e variantes do sistema CRISPR-Cas9 centráronse en maximizar o control no seu uso. Concretamente, preténdese mellorar a súa especificidade, eficiencia e granularidade do seu poder de edición. As técnicas poden ser subdivididas e clasificadas polo compoñente do sistema que modifican. Estas inclúen o uso de diferentes variantes ou novas creacións da proteína Cas, o uso dunha proteína efectora totalmente diferente, a modificación do ARNgs ou o uso dunha estratexia algorítmica para identificar as solucións óptimas.
A especificidade é un importante aspecto para mellorar o sistema CRISPR-Cas9 porque os efectos fóra de diana que xera teñen graves consecuencias para o xenoma da célula e aconsellan precaución no seu uso. Minimizar os efectos fóra de diana é así maximizar a seguridade do sistema. Novas variacións das proteínas Cas9 que incrementan a especificidade inclúen as proteínas efectoras con eficiencia e especificidade comparables ás da SpCas9 orixinal, que poden dirixirse a secuencias diana previamente non alcanzables, e unha variante que virtualmente non ten mutacións fóra de diana.[87][88] Tamén se investigou a preparación por enxeñaría de novas proteínas Cas9, incluíndo algunhas que substitúen parcialmente ribonucleótidos en ARNcr por desoxirribonucleótidos (de ADN), e un mutante de Cas9 guiado a estrutura, todos os cales teñen efectos fóra de diana reducidos.[89][90] Os ARNgs repetidamente truncados e os ARNg moi estabilizados tamén diminúen os efectos fóra de diana.[91][92] Os métodos computacionais incluíndo a aprendizaxe de máquina utilizáronse para predicir a afinidade e crear secuencias únicas para o sisema para maximizar a especificidade para unha diana dada.[93][94]
Varias variantes de CRISPR-Cas9 permiten a activación de xenes ou edición do xenoma cun causante externo como a luz ou pequenas moléculas.[95][96][97] Estes inclúen sistemas CRISPR fotoactivables desenvolvidos fusionando proteínas sensibles á luz cun dominio activador e unha dCas9 para a activación xénica,[98][99] ou fusionar dominios sensibles á luz similares con dous construtos de split-Cas9,[100][101] ou incorporando aminoácidos non naturais engaiolados á Cas9,[102] ou modificando os ARNs guías con complementos fotoactivables para a edición do xenoma.[103]
Entre os métodos de control da edición do xenoma con pequenas moléculas están unha Cas9 alostérica, sen ningunha edición de fondo detectable, que activa a unión e corte coa adición de 4-hidroxitamoxifén (4-HT),[95] unha Cas9 ligada a inteína sensible a 4-HT,[104] ou unha Cas9 que é sensible a 4-HT cando se fusiona a catro dominios ERT2.[105] A split-Cas9 inducible por inteína permite a dimerización de fragmentos de Cas9[106] e o sistema de split-Cas9 inducible por rapamicina foi desenvolvido fusionando dous construtos de split-Cas9 con fragmentos de FRB e FKBP.[107] Outros estudos conseguiron inducir a transcrición de Cas9 cunha pequena molécula, a doxiciclina.[108][109] Tamén se poden usar pequenas moléculas para mellorar a reparación dirixida por homoloxía,[110] a miúdo inhibindo a vía da unión de extremos non homólogos e/ou a vía da unión de extremos mediada por theta.[111][112] Creouse un sisema coa proteína efectora Cpf1 que é inducido polas pequenas moléculas VE-822 e AZD-7762.[113] Estes sistemas permiten o control condicional da actividade de CRISPR para mellorar a precisión, eficiencia e control espazotemporal. O control espazotemporal é unha forma de eliminar os efectos fóra de diana, xa que só certas células ou partes do organismo necesitan ser modificadas, e así poden utilizarse a luz ou pequenas moléculas como modo de realizalo. A eficiencia do sistema CRISPR-Cas9 aumentouse tamén grandemente coa entrega axeitada de instrucións do ADN para crear as proteínas e reactivos necesarios.[113]
O CRISPR tamén utiliza proteínas editoras dun só par de bases para crear edicións específicas nunha ou dúas bases da secuencia diana. Fusionouse CRISPR/Cas9 con encimas específicos que inicialmente só podían cambiar mutacións de C por T e G por A e as súas inversas. Isto realizouse finalmente sen necesidade de ningún corte no ADN.[114][115][116] Coa fusión doutro encima, o sistema CRISPR-Cas9 de edición de bases pode tamén editar C por G e o seu inverso.[117]
As repeticións palindrómicas curtas agrupadas espazadas regularmente (CRISPR, do inglés clustered regularly interspaced short palindrome repeats) do sistema CRISPR/Cas9 son unha tecnoloxía de edición de xenes que pode inducir roturas de dobre febra en calquera punto no que se poidan unir ácidos ribonucleicos guías (ARNg) coa secuencia PAM (motivo adxacente protoespazador, en inglés protospacer adjacent motif).[118] Os mutantes de sitio activo de Cas9 poden inducir tamén amosegas nunha soa febra,[119] tamén coñecidos como nickases Cas9.[120] Simplemente cambiando a secuencia do ARNg, a endonuclease Cas9 pode entregarse a un xene de interese e inducir roturas de dobre febra.[121] A eficiencia da endonuclease Cas9 e a facilidade coa cal se poden alcanzar os xenes diana levou ao desenvolvemento de bibliotecas de CRISPR-knockout (KO) tanto en células de ratos coma humanas, as cales poden cubrir conxuntos de xenes específicos de interese ou mesmo o xenoma completo.[122][123] O cribado CRISPR axudou a crear unha perturbación xenética sistemática e de alto rendemento en organismos modelo. Esta perturbación xnética é necesaria para comprender a función do xene e a súa regulación epixenética.[124] A vantaxe das librarías CRISPR de pools é que poden alcanzarse máis xenes diana á vez.
As bibliotecas knock-out créanse de maneira que teñan igual representación e actuación con todos os ARNg e porten un marcador de selección fluorescente ou antibiótico que pode utilizarse para recuperar células transducidas.[118] Hai dous sistemas de plásmidos nas bibliotecas CRISPR/Cas9. No primeiro está todo nun plásmido, no que o ARNgs e o Cas9 son producidos simultaneamente nunha célula transfectada. O segundo é un sistema de dous vectores: o ARNgs e os plásmidos Cas9 son entregados separadamente.[124] É importante entregar miles de vectores que conteñen ARNgs únicos nun só recipiente de células por transdución viral a baixa multiplicidade de infección (tipicamente de 0,1–0,6), porque prevén a probabilidade de que un clon de células individual tome máis dun tipo de ARNgs, xa que doutro modo isto pode levar a unha asignación incorrecta de xenotipo a fenotipo.[122]
Unha vez que se prepara a bibliotecas de pools é necesario levar a cabo unha secuenciación profunda (secuenciación de seguinte xeración) de ADN de plásmido amplificado por PCR para revelar a abundancia de ARNgs. As células de interese poden ser infectadas consecuentemente pola biblioteca e despois seleccionadas de acordo co fenotipo. Hai dous tipos de selección: negativa e positiva. Por selección negativa detéctanse eficientemente as células mortas ou de lento crecemento. Poden identificar os xenes esenciais de supervivencia, que poden servir como candidatos para fármacos dirixidos a diana molecularmente. Por outra parte, a selección positiva dá unha conxunto de poboacións que adquiriron vantaxes de crecemento por mutaxénese aleatoria.[118] Despois da selección o ADN xenómico é recollido e secuenciado por secuenciación de seguinte xeración. O empobrecemento ou enriquecemento de ARNgs detéctase e compárase coa biblioteca de ARNgs orixinal, anotada co xene diana ao que corresponda o ARNgs. Despois, a análise estatística identifica xenes que é significativamente probable que sexan relevantes para o fenotipo de interese.[122]
| Biblioteca | ID | Especie | PI | Xenes diana | Número de ARNg por xene | ARNg totais |
|---|---|---|---|---|---|---|
| Biblioteca de CRISPR knockout de rato Bassik | 1000000121–1000000130 | Rato | Bassik | Varía (~23.000 en total) | ~10 | Varía |
| Biblioteca de CRISPR knockout do xene supresor de tumores de rato | 113584 EFS backbone 113585 TBG backbone | Rato | Chen | 56 | ~4 | 286 |
| Biblioteca de todo o xenoma de ratos Brie | 73632 (1 plásmido) 73633 (2 plásmido) | Rato | Doench and Root | 19.674 | 4 | 78.637 |
| Biblioteca de CRISPR knockout humana Bassik | 101926–101934 | Humanos | Bassik | Varía (~20.500 en total) | ~10 | Varía |
| Biblioteca de todo o xenoma humana Brunello | 73179 (1 plásmido) 73178 (2 plásmidos) | Humanos | Doench and Root | 19.114 | 4 | 76.441 |
| Biblioteca de knockout de todo o xenoma mini-humana baseada en AsCpf1 | 130630 | Humanos | Draetta | 16.977 | 3–4 | 17.032 matrices |
Á parte das bibliotecas de knockout hai tamén bibliotecas de knock-down (CRISPRi) e de activación (CRISPRa), que usan a capacidade das Cas9-proteínas de fusión desactivadas proteoliticamente (dCas9) de unirse ao ADN diana, o que significa que o xene de interese non é cortado senón que é sobreexpresado ou reprimido. Isto fai ao sistema CRISPR/Cas9 aínda máis interesante para a edición de xenes. A proteína dCas9 inactiva modula a expresión xénica ao dirixir represores ou activadores de dCas9 cara a sitios de comezo da transcrición ou promotores dos xenes diana. Para reprimir xenes, Cas9 pode fusionarse ao dominio efector KRAB que fai un complexo co ARNg, mentres que CRISPRa utiliza dCas9 fusionada a diferentes dominios de activación transcricional, que son ademais dirixidos polo ARNg a rexións promotoras para regular á alza a expresión.[126][127][128]
A modificación xenética por Cas9 permite unha xeración rápida e eficiente de modelos transxénicos dentro do campo da xenética. Cas9 pode ser facilmente introducido nas células diana xunto co ARNgs por medio de transfección de plásmidos para modelizar o espallamento de enfermidades, a resposta das células e a defensa contra a infección.[129] A capacidade de Cas9 para ser introducida in vivo permite a creación de modelos máis axeitados da función xénica e efectos das mutacións, mentres que se evitan a mutacións fóra de diana observadas tipicamente nos métodos máis vellos de enxeñaria xenética.
A revolución que supuxo CRISPR e Cas9 nos modelos xenómicos non afecta só os mamíferos. Os modelos xenómicos tradicionais como Drosophila melanogaster, un dos primeiros organismos modelo, fixéronse máis requintados na súa resolución co uso de Cas9.[129] Cas9 usa promotores específicos de célula, o que permite un uso controlado de Cas9. O método da Cas9 é axeitado para tratar doenzas debido a que a acción do encima Cas9 só afecta a certos tipos celulares. As células que sofren a terapia Cas9 poden tamén ser extraídas e reintroducidas para proporcionar efectos amplificados da terapia.[130]
CRISPR-Cas9 pode utilizarse para editar o ADN de organismos in vivo e para eliminar xenes concretos ou mesmo cromosomas enteiros dun organismo en calquera momento do seu desenvolvemento. Entre os cromosomas que foron eliminados con éxito in vivo usando as técnicas CRISPR están o cromosoma Y e o X de ratos de laboratorio adultos e os cromosomas humanos 14 e 21; o primeiro en liñas de células nai embrionarias de ratos aneuploides cun cromosoma humano extra, e o segundo en células nais pluripotentes inducidas humanas. Este método podería ser útil para o tratamento de trastornos xenéticos causados por ter un número anormal de cromosomas, como a síndrome de Down e os trastornos intersexo.[131]
A edición do xenoma in vivo usando CRISPR-Cas9 realizouse con éxito en numerosos organismos modelo, incluíndo Escherichia coli,[132] Saccharomyces cerevisiae,[133] [134]Candida albicans, Methanosarcina acetivorans,[135][136] Caenorhabditis elegans,[137] Arabidopsis spp.,[138] Danio rerio,[139] e Mus musculus.[140][141] Outros éxitos conseguíronse no estudo da bioloxía básica, na creación de modelos de enfermidades,[137][142] e no tratamento experimental de modelos de enfermidades.[143]
Un problema é que os efectos da edición do xenoma fóra de diana (edición de xenes ao lado dos que realmente se pretendía) pode confundir os resultados dun experimento de edición de xenes por CRISPR, é dicir, o cambio fenotípico observado pode non deberse á modificación do xene diana, senón á dalgún outro xene. Fixéronse modificacións ao CRISPR para minimizar a posibilidade de efectos fóra de diana. Adoitan recomendarse experimentos de CRISPR ortogonal para confirmar os resultados dun experimento de edición de xenes.[144][145]
CRISPR simplifica a creación de organismos modificados xeneticamente para a investigación que imitan unha enfermidade ou mostran o que ocorre cando un xene é sometido a knock-down ou mutado. A CRISPR pode utilizarse na liña xerminal para crear organismos nos cales o xene diana queda cambiado en todas partes onde se encontre (é dicir, en todas as células/tecidos/órganos dun organismo multicelular), ou pode utilizarse en células non xerminais para crear cambios locais que só afectan a certas poboacións celulares do organismo.[146][147][148]
CRISPR pode utilizarse para crear modelos celulares humanos de enfermidades.[149] Por exemplo, cando se aplica a células nais pluripotentes humanas, CRISPR utilizouse para introducir mutacións dirixidas a diana en xenes relevantes para a enfermidade renal poliquística e a glomeruloesclerose segmentaria e focal.[150] Estas células nai pluripotentes modificadas por CRISPR fixéronse despois crecer en organoides de ril humano que mostraron fenotipos específicos de enfermidades. Os organoides de ril formados a partir de células nais con mutacións para a enfermidade renal poliquística formaron estruturas quísticas translúcidas grandes a partir dos túbulos renais. Os quistes podían acadar dimensións macroscópicas, de ata un centímetro de diámetro.[151] Os organoides de ril con mutacións nun xene ligadas á glomeruloesclerose segmentaria e focal desenvolveron defectos nas unións celulares entre os podocitos, que son as células filtradoras que están afectadas nesa doenza. Isto debíase á incapacidade dos podocitos de formar microvilosidades entre as células adxacentes.[152] Un dato importante é que estes fenotipos de enfermidades estaban ausentes nos organoides control de idéntico fondo xenético, pero que carecían de modificacións CRISPR.[150]
Seguiuse unha estratexia similar para modelizar a síndrome do QT longo en cardiomiocitos derivados de células nai pluripotentes.[153] Estes modelos celulares xerados por CRISPR, con controis isoxénicos, proporcionan un novo modo de estudar as doenzas humanas e de ensaiar fármacos.
A tecnoloxía CRISPR-Cas propúxose como tratamento para múltiples doenzas humanas, especialmente as que teñen unha causa xenética.[154] A súa capacidade de modificar secuencias específicas de ADN convértea nunha ferramenta coa potencial capacidade de arranxar mutacións que causan enfermidades. As primeiras investigacións sobre isto en modelos animais indican que as terapias baseadas na tecnoloxía CRISPR poderían utilizarse para tratar unha ampla gama de doenzas,[155] incluíndo o cancro,[156] a proxeria,[157] a beta-talasemia,[158][159][160] a anemia falciforme,[160][161] a hemofilia,[162] a fibrose quística,[163] a distrofia muscular de Duchenne,[164] a enfermidade de Huntington,[165][166] a amiloidose de transtiretina[41] e doenzas cardíacas.[167] A CRISPR tamén se utilizou para previr a malaria eliminando mosquitos, o cal podería eliminar o vector e a enfermidade en humanos.[168] A CRISPR pode tamén ter aplicacións na enxeñaría de tecidos e na medicina rexenerativa, como crear vasos sanguíneos humanos que carezan da expresión das proteínas MHC de clase II, que a miúdo causan o rexeitamento de transplantes.[169]
Ademais, os ensaios clínicos para curar a beta-talasemia e a anemia falciforme en pacientes humanos usando a tecnoloxía CRISPR-Cas9 tiveron resultados prometedores.[170][171]
Non obstante, seguen existindo algunhas limitacións no uso desta tecnoloxía en terapia xénica, como son: a frecuencia relativamente alta de efectos fóra de diana, a necesidade dunha secuencia PAM preto do sitio diana, a apoptose mediada por p53 por roturas de dobe febra inducidas por CRISPR e a toxicidade inmunoxénica debida ao sistema de entrega que normalmente é un virus.[172]
O tratamento CRISPR para a LCA10 (a variante máis común da amaurose conxénita de Leber, que é a principal causa de cegueira herdada na nenez) modifica o xene de fotorreceptor defectuoso do paciente.
En marzo de 2020 nun estudo sobre esta doenza nos Estados Unidos, patrocinado por Editas Medicine, un primeiro paciente voluntario recibiu unha dose do tratamento para testar a súa seguiridade. En xuño de 2021 empezou o recrutamento de participantes para unha dose adulta alta e unha cohorte pediátrica con 4 pacientes voluntarios en cada unha. A administración de doses ás novas cohortes esperábase que estivese completa en xullo de 2022.[173] En novembro de 2022, Editas informou que o 20% dos pacientes tratados tiveran melloramentos importantes, pero tamén anunciou que a poboación diana resultante era demasiado pequena como para soportar un desenvolvemento continuado independente.[174]
A CRISPR tamén encontrou moitas aplicacións no desenvolvemento de inmunoterapias baseadas en células.[175] O primeiro ensaio clínico con CRISPR empezou en 2016. Consistiu en extraer células inmunitarias de persoas con cancro de pulmón, usar CRISPR para editar o xene expresado PD-1, despois administrar as células alteradas de volta á mesma persoa. Outros vinte ensaios estaban en marcha ou case listos, principalmente na China en 2017.[156]
En 2016 a FDA dos Estados Unidos aprobou un ensaio clínico con CRISPR no que se extraerían células T de persoas con diferentes tipos de cancro e despois se lles administrarían ás mesmas persoas esas células unha vez modificadas por enxeñaría.[176]
En novembro de 2020 en modelos animais de rato, utilizouse CRISPR para tratar o glioblastoma (tumor cerebral de rápido crecemento) e o cancro de ovario metastático, xa que eses son dous cancros con peor prognóstico e diagnostícanse normalmente en estadios avanzados. Os tratamentos orixinaron unha inhibición do crecemento do tumor, un incremento da supervivencia do 80% no caso do cancro ovárico metastático e a apoptose da célula tumoral, e no caso do glioblastoma unha inhibición do crecemento do tumor dun 50% e unha mellora da supervivencia do 30%.[177]
En outubro de 2021 CRISPR Therapeutics anunciou os resultados do seu ensaio en fase 1 en marcha nos Estados Unidos para unha terapia de célula T aloxénica. Estas células tomáronse de doantes sans e foron editadas para atacar células cancerosas e evitar ser vistas como unha ameaza polo sistema inmunitario do receptor, e despois multiplicáronse en enormes lotes que podían administrarse a un gran número de pacientes.[173]
En decembro de 2022 unha rapaza británica de 13 anos que fora diagnosticada de leucemia linfoblástica aguda de células T incurable foi curada no Great Ormond Street Hospital, no primeiro uso documentado de edición de xenes terapéutica para este propósito, despois de someterse a seis meses de tratamento experimental, cando outros tipos de tratamento fallaran previamente. O procedemento incluía reprogramar células T sas para destruír células T cancerosas para en primeiro lugar eliminar a leucemia, e despois reconstruír o sistema inmunitario da paciente desde cero usando células inmunitarias sas.[178] O equipo utilizou edición BASE e tratara previamente un caso de leucemia linfoblástica aguda en 2015 usando TALENs.[179]
A diabetes tipo 1 é un trastorno endócrino que é o resultado da perda de células beta pancreáticas que producen insulina, unha hormona vital para que o azucre do sangue entre na células para producir enerxía. Os investigadores estiveron intentando o transplante de células beta sas. Utilizouse CRISPR para editar as células para reducir as posibilidades de que o corpo do paciente rexeite o transplante. En febreiro de 2022 realizouse un ensaio en fase 1 na cal recibiu o tratamento un paciente voluntario.[173][180]
O virus da inmunodeficiencia humana (VIH) é un virus que ataca o sistema inmunitario do corpo. Aínda que existen tratamentos efectivos para manter a raia a enfermidade e que permiten aos pacientes ter unha vida sa, o VIH é un retrovirus, o que significa que integra unha versión inactiva del no xenoma humano, que se pode activar en certos momentos. A CRISPR pode utilizarse para eliminar selectivamente o virus do xenoma deseñando un ARN guía que teña como diana o xenoma integrado do VIH. Un probelma con esta estratexia é que require a eliminación do xenoma integrado do VIH de case todas as células, o cal, sendo realista, pode ser difícil de conseguir.[173]
As "nucleases guiadas por ARN" baseadas en CRISPR-Cas poden utilizarse para alterar factores de virulencia, xenes que codifican resistencia a antibióticos e outras secuencias medicamenrte relevantes de interese. Esta tecnoloxía representa unha nova forma de terapia antimicrobiana e unha estratexia para manipular poboacións bacterianas.[181][182] Estudos recentes indican unha correlación entre a interferencia do locus CRISPR-Cas e a adquisición e resistencia a antibióticos.[183] Este sistema proporciona protección á bacteria ante ADN alleo invasor, como o de transposóns, bacteriófagos e plásmidos. Este sistema exerce unha forte presión de selección para a adquisición de resistencia a antibióticos e factores de virulancia en patóxenos bacterianos.[183]
As terapias baseadas na tecnoloxía de edición de xenes por CRISPR–Cas3 entregadas por bacteriófagos preparados por enxeñaría poderían usarse para destruír o ADN diana en patóxenos.[184] Cas3 é máis destrutuiva que a máis coñecida Cas9.[185][186]
Diversas investigacións suxiren que CRISPR é un modo efectivo de limitar a replicación de múltiples herpesvirus. Conseguiu erradicar o ADN viral no caso do virus de Epstein–Barr (EBV). O uso de CRISPRs antiherpesvirus ten prometedoras aplicacións, como eliminar o EBV causante de cancro das células tumorais, axudando a eliminar os invasores víricos en órganos de doantes para pacientes inmunocomprometidos, ou impedir brotes de Herpes labialis e infeccións recorrentes no ollo ao bloquear a reactivación do HSV-1. En agosto de 2016 estes procedementos estaban esperando a ser testados.[187]
Os resultados iniciais no tratamento e cura do VIH foron bastante positivos, en outubro de 2021 9 dun total de 23 ratos humanizados tratados cunha combinación de anti-retrovirais e CRISPR/Cas-9 tiñan o virus indetectable, mesmo despois do usual período de rebote. Ningún dos dous tratamentos por si só tiña ese efecto.[188] Os ensaios clínicos en humanos empezaron en 2022.[189][190]
A CRISPR pode facer revivir o concepto de xenotransplante de órganos animais a persoas. Os retrovirus presentes en xenomas animais podía causar dano aos receptores do transplante. En 2015 un equipo eliminou 62 copias dunha determinada secuencia de ADN retroviral do xenoma do porco nunha célula epitelial de ril.[191] Demostrouse recentemente por primeira vez a capacidade de que nazan vivos espécimes de porcos despois de eliminar eses retrovirus dos xenomas dos embrións usando CRISPR.[192]
A CRISPR-Cas9 pode utilizarse para investigar e identificar as diferenzas xenéticas entre humanos e outros primates, como os simios, especialmente no cerebro. Por exemplo, ao reintroducir variantes de xenes arcaicos en organoides de cerebro para mostrar unha influencia na neuroxénese,[193] a lonxitude da metafase de proxenitores apicais do neocórtex en desenvolvemento,[194] ou polo knockout dun xene en células nais embrionarias para identificar un regulador xenético que por transicións da forma da célula temperás conduciron á expansión evolutiva do prosencéfalo humano.[195][196] Un estudo describiu unha influencia importante dunha variante dun xene arcaico no neurodesenvolvemento[197][198] que pode ser un artefacto dun efecto colateral do CRISPR,[199][200] xa que non puido ser replicado nun estudo posterior.[201]
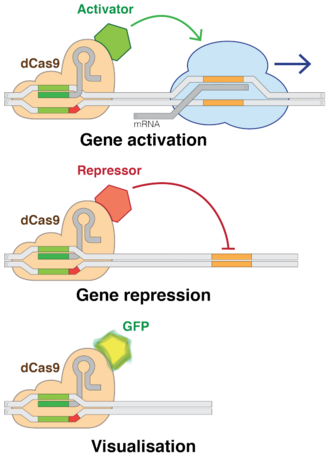
O uso de versións "mortas" de Cas9 (dCas9) elimina a capacidade de cortar o ADN de CRISPR, mentres que conserva esa capacidade para actuar sobre as secuencias desexadas. Múltiples grupos de investigadores engadiron varios factores reguladores a dCas9s, permitíndolles activar ou desactivar case calquera xene ou axustar o seu nivel de actividade.[191] Igual que a interferencia de ARN (RNAi), a interferencia por CRISPR (CRISPRi) desactiva xenes de forma reversible ao unirse á diana pero non cortar o sitio. O sitio diana é metilado, modificando así epixeneticamente o xene. Esta modificación inhibe a transcrición. Estas modificacións situadas con precisión poden usarse despois para regular os efectos na expresión xénica da dinámica do ADN despois da inhibición de certas secuencias xenómicas do ADN. Nos últimos anos, investigáronse intensamente as marcas epixenéticas en diferentes células humanas e atoparon que certos padróns nas marcas se correlacionaban con todas as cousas desde o crecemento do tumor á actividade cerebral.[10] Inversamente, a activación mediada por CRISPR (CRISPRa) promove a transcrición xénica.[202] Cas9 é unha forma efectiva de actuar sobre unha diana e silenciar xenes específicos a nivel do ADN.[203] En bacterias, a presenza de Cas9 soa é dabondo para bloquear a transcrición. Para aplicacións en mamíferos, engádese unha sección da proteína. O seu ARN guía únese a secuencias do ADN regulatorias chamadas promotores que preceden inmediatamente ao xene diana.[204]
Cas9 utilizouse para transportar factores de transcrición sintéticos que activaban xenes humanos específicos. A técnica conseguiu un forte efecto ao dirixir múltiples construtos CRISPR a localizacións lixeiramente diferentes do promotor do xene.[204]
En 2016 os investigadores demostraron que o CRISPR procedente dunha bacteria común da boca podería usarse para editar o ARN. Os investigadores buscan en bases de datos que conteñen centos de millóns de secuencias xenéticas aquelas que lembran os xenes CRISPR. Estudaron a fusobacteria Leptotrichia shahii, a cal ten un grupo de xenes que lembran os xenes CRISPR, pero con importantes diferenzas. Cando os investigadores equiparon outras bacterias con eses xenes, aos cales chamaron C2c2, atoparon que os organismos adquirían unha nova defensa.[205] O C2c2 foi despois renomeado como Cas13a para axustarse á nomenclatura estándar dos xenes Cas.[206]
Moitos virus codifican a súa información xenética no ARN en vez de no ADN, que eles reprograman para facer novos virus. O VIH e o poliovirus son virus dese tipo. As bacterias con Cas13 producen moléculas que poden desfacer o ARN, destruíndo o virus. Preparar convenientemente estes xenes serve para editar calquera molécula de ARN.[205]
Os sistemas CRISPR-Cas poden tamén epregarse para editar xenes de microARN e ARN non codificante longo en plantas.[207]
As edicións dirixidas para corrixir secuencias mutadas foi proposta por primeira vez e demostrada en 1995.[208] Este traballo inicial usou oligonucleótidos de ARN antisentido sintético complementario dunha mutación de codón de stop premaduro nunha secuencia de distrofina para activar a edición de adenina (A) a inosina (I) do codón de stop xerando un codón que se pode ler nun sistema modelo de células de Xenopus.[208] Aínda que isto tamén causou transicións inadvertidas de A a I en lugares próximos, as transicións de A a I (lidas como G) poden corrixir os tres posibles codóns de stop, pero non poden crear un novo codón de stop. Por tanto, os cambios causaron >25 % de corrección do codón de stop diana con lectura dunha secuencia augas abaixo de luciferase reporteira. Posteriores traballos feitos por Rosenthal conseguiron editar unha secuencia de ARNm mutada en cultivos de células de mamíferos ao dirixir un oligonucleótido ligado á citidina desaminase para corrixir unha secuencia mutada de fibrose quística.[209] Máis recentemente, empregouse CRISPR-Cas13 fusionada a desaminases para dirixir a edición de ARNm.[210]
En 2022 informouse do uso de Cas7-11, máis apropiada para a edición de ARN terapéutica que Cas13.[211][212] Permite cortes suficientemente ben dirixidos a diana e unha verisón inicial xa foi utilizada para a edición in vitro en 2021.[213]
A diferenza da edición de ADN, que é permanente, os efectos da edición do ARN, incluíndo as posibles mutacións fóra de diana no ARN, son transitorias e non se herdan. A edición do ARN é, por tanto, considerada menos perigosa. Ademais, pode soamente necesitar un ARN guía ao usar a proteína ADAR xa atopada en células humanas e de moitos outros eucariotas en vez de ter que introducir unha proteína allea no corpo.[214]
A xenética dirixida (gene drive) pode proporcionar unha potente feramenta para restablecer o equilibrio dos ecosistemas ao eliminar especies invasoras. Hai dúbidas sobre a súa eficacia, e sinaláronse posibles consecuencias inesperadas nas especies diana así como nas especies non diana, especialmente pola posibilidade dunha liberación accidental dos laboratorios á natureza. Propuxéronse varias salvagardas para asegurar a contención da xenética dirixida experimental de tipo molecular, reprodutivo e ecolóxico.[215] Moitos recomendan que a inmunización e a xenética dirixida inversa sexan desenvolvidos en tándem coa xenética dirixida para sobreescribir os seus efectos se for necesario.[216] Hai consenso en que os efectos a longo prazo deben ser estudados máis detalladamente ante a posibilidade de alteracións ecolóxicas que non poidan ser corrixidas con xenética dirixida inversa.[217]
As bibliotecas de secuenciación empobrecidas a miúdo teñen abundantes secuencias non desexadas. Cas9 pode empobrecer especificamente as secuencias non desexadas con roturas de dobre febra con ata un 99 % de eficiencia e sen efectos fóra de diana significativos, como se viu con encimas de restrición. O tratamento con Cas9 pode empobrecer o ARNr abundante, mentres que incrementa a sensibilidade do patóxeno en bibliotecas de RNA-seq.[218]
A edición epixenómica ou enxeñaría epixenómica é un tipo de enxeñaría xenética na cal se modifica o epixenoma en sitios específicos usando moléculas preparadas por enxeñaría dirixidas a ditos sitios (en oposición ás modificacións en todo o xenoma). Mentres que a edición de xenes implica cambiar a propia secuencia de ADN, a edición epixenética só supón a modificación e presentación de secuencias de ADN a proteínas e outros factores que se unen ao ADN que inflúen na función do ADN. Ao "editar” características epixenómicas deste xeito, pódese determinar o papel biolóxico exacto dunha modificación epixenética no sitio en cuestión.
As proteínas preparadas por enxeñaría usadas para a edición epixenómica están compostas dun dominio de unión ao ADN que ten como diana secuencias específicas e un dominio efector que modifica características epixenómicas. Actualmente, están utilizándose predominantemente tres grandes grupos de proteínas que se unen ao ADN para a edición epixenómica, que son: proteínas de dedo de cinc, efectores similares a activadores de transcrición (TALEs) e fusións de Cas9 deficientes en nuclease (CRISPR).
A regulación dirixida de xenes relacionados con enfermidades podería permitir novas terapias para moitas doenzas, especialmente en casos nos que as terapias xénicas adecuadas non están aínda desenvolvidas ou son inapropiadas.[219] Aínda que as consecuencias a nivel transxeneracional e de poboación non se comprenden totalmente, pode converterse nunha importante ferramenta para aplicala á xenómica funcional e á medicina personalizada.[220] Igual que na edición de ARN, non implica cambios xenéticos e os riscos que os acompañan.[219] Un exemplo dun posible uso funcional da edición do epixenoma describiuse en 2021: a represión da expresión xénica de Nav1.7 por CRISPR-dCas9, que mostrou un potencial terapéutico en tres modelos de ratos de dor crónica.[221][222]
En 2022 fixeronse investigacións que avaliaron a súa utilidade na redución dos niveis de proteína tau, regulando unha proteína que intervén na enfermidade de Huntington, para tratar unha forma herdada de obesidade, e a síndrome de Dravet.[223]
A combinación de CRISPR-Cas9 con integrases orixinou unha técnica para facer grandes edicións sen as problemáticas roturas de dobre febra, como se demostrou con PASTE[224] en 2022. Informouse que se podía usar para entregar xenes de ata 36.000 pares de bases de ADN a varios tipos de células humanas e podería usarse para tratar doenzas causadas por un gran número de mutacións.[225][226]
A edición de calidade (prime editing en inglés)[227] ou edición de bases é un refinamento da CRISPR para poder inserir ou eliminar con exactitude seccións do ADN. As edicións de CRISPR non son sempre perfectas e os cortes poden acabar aparecendo no sitio incorrecto. Ambas cuestións son un problema á hora de usar esta tecnoloxía en medicina.[228] Esta edición de calidade non corta o ADN bicatenario senón que utiliza o aparato de busca de diana da CRISPR para transportar un encima adicional á secuencia desexada, onde este converte un só nucleótido noutro con precisión.[229] A nova molécula guía, chamada ARN guía de prime editing (pegRNA en inglés), contén un molde de ARN para que se engada unha nova secuencia de ADN ao xenoma na localización onde está a diana. Para isto cómpre unha segunda proteína, unida a Cas9: un encima transcriptase inversa, que pode facer unha nova febra de ADN a partir do molde de ARN e inserila no sitio onde se fixo a amosega.[230] Eses tres eventos emparellados independentemente proporcionan cada un unha oportunidade para impedir eventos fóra de secuencia, o cal incrementa significativamente a flexibilidade á hora de dirixirse á diana e a precisión da edición.[229] A prime editing foi desenvolvida por investigadores do Broad Institute da MIT e Harvard en Massachusetts.[231] Serán necesarios máis traballos para optimizar os métodos.[231][230]
En marzo de 2015 múltiples grupos de investigadores anunciaran que estaban realizando investigacións coa intención de establecer os fundamentos para aplicar a CRISPR a embrións humanos para a enxeñaría da liña xerminal humana, incluíndo laboratorios dos Estados Unidos, a China e o Reino Unido, así como a compañía biotecnolóxica estadounidense OvaScience.[232] Os científicos, entre eles o codescubridor do CRISPR, instaron unha moratoria mundial na aplicación do CRISPR á liña xerminal humana, especialmente para o uso clínico. Dixeron que "os científicos deberían evitar incluso o intento, en xurisdicións permisivas, da modificación do xenoma na liña xerminal para a aplicación clínica en humanos" ata que todas as implicacións "se discutan entre os científicos e as organizacións gobernamentais".[233][234] Estes científicos apoiaron unha maior investigación a baixo nivel sobre CRISPR e non consideralo como un método suficientemente desenvolvido de uso clínico para facer cambios herdables en seres humanos.[235]
En abril de 2015 científicos chineses informaron dos resultados dun intento de alterar o ADN de embrións humanos non viables usando CRISPR para corrixir unha mutación que causa a beta-talasemia, un trastorno herdable que pode ser mortal.[236][237] O estudo fora previamente rexeitado polas revistas Nature e Science en parte debido a consideracións éticas.[238] Os experimentos tiveron como resultado o cambio con éxito de só algúns dos xenes pretendidos, e presentaban efectos fóra de diana sobre outros xenes. Os investigadores afirmaron que o CRISPR non está listo para a aplicación clínica en medicina reprodutiva.[238] En abril de 2016 informouse que científicos chineses fixeran un segundo intento fracasado de alterar o ADN de embrións humanos non viables usando CRISPR; esta vez para alterar o xene CCR5 para facer que o embrión fose resistente á infección por VIH.[239]
En decembro de 2015 un Cumio Internacional sobre a Edición de Xenes Humanos tivo lugar en Washington baixo a dirección de David Baltimore. Membros das academias científicas nacionais dos Estados Unidos, o Reino Unido e a China discutiron os aspectos éticos da modificación da liña xerminal. Estiveron de acordo en apoiar a investigación básica e clínica baixo certas directrices legais e éticas. Fíxose unha distinción específica entre as células somáticas, nas que os efectos das edicións están limitadas a un só individuo, e as células da liña xerminal, nas que os cambios no xenoma poden herdarse polos descendentes. As modificacións herdables poderían ter consecuencias non pretendidas e de longo alcance para a evolución humana, tanto xeneticamente (por exemplo, as interaccións xene–ambiente) coma culturalmente (por exemplo, o darwinismo social). A alteración dos gametocitos e embrións para xerar cambios herdables en humanos definiuse como irresponsable. O grupo concordou en iniciar un foro internacional para tratar eses asuntos e harmonizar as regulacións entre os países.[240]
En febreiro de 2017 o Comité das Academias Nacionais de Ciencias, Enxeñaría e Medicina dos Estados Unidos (NASEM) sobre a Edición de Xenes Humanos publicou un informe revisando as cuestións éticas, legais e científicas da tecnoloxía de enxeñaria xenómica. A conclusión do informe afirmaba que a edición xenómica herdable non é permisible agora, pero podería ser xustificable para certas condicións médicas, pero non xustificaban o uso de CRISPR para a mellora das características humanas.[241]
En novembro de 2018 Jiankui He na China anunciou que editara dous embrións humanos para intentar desactivar o xene da CCR5, que codifica un receptor que usa o VIH para entrar nas células. Dixo que unhas poucas semanas antes naceran dúas nenas xemelgas tratadas para facelas invulnerables ao VIH, chamadas Lulu e Nana, pero que as nenas aínda portaban copias funcionais do CCR5 xunto con CCR5 desactivado (un caso de mosaicismo) e eran aínda vulnerables ao VIH. O traballo foi amplamente condenado como falto de ética, perigoso e prematuro.[242] Un grupo internacional de científicos solicitou unha moratoria mundial sobre a edición xenética de embrións humanos.[243]
Os principios regulatorios para o sistema CRISPR-Cas9 varían arredor do mundo. En febreiro de 2016 científicos británicos recibiron permiso dos reguladores para modificar xeneticamente embrións humanos usando CRISPR-Cas9 e técnicas relacionadas. Porén, aos investigadores prohibíuselles implantar os embrións e ditos embrións foron destruídos sete días despois.[244]
Os Estados Unidos teñen un elaborarado sistema regulatorio interdepartmental para avaliar novos alimentos modificados xeneticamente. Por exemplo, a Lei de Protección de Riscos para a Agricultura de 2000 dá ao Departamento de Agricultura dos Estados Unidos a autoridade de supervisar a detección, control, erradicación, supresión, prevención ou demora do espallamento de pragas agrícolas ou malas herbas prexudiciais para protexer a agricultura, o medio ambiente e a economía dos Estados Unidos. A lei regula calquera organismo modificado xeneticamente que utilice o xenoma dunha "praga agrícola" predefinida ou calquera planta non previamente categorizada.[245] En 2015 Yinong Yang conseguiu desactivar 16 xenes específicos no fungo Agaricus bisporus (champiñón común) para facer que non se volvese marrón. Como non engadiran ningún ADN de especie allea (transxénico) a este organismo, o fungo non podía ser regulado polo USDA baixo a Section 340.2.[246] O champiñón de Yang foi o primeiro organismo modificado xeneticamente co sistema de proteínas de CRISPR-Cas9 que pasou a regulación dos Estados Unidos.[247]
En 2016 o USDA patrocinou un comité para considerar futuras políticas regulatorias para as técnicas de modificación xenética por vir. Coa axuda das Academias Nacionais de Ciencias, Enxeñaria e Medicina dos Estados Unidos, os grupos con interese especial xuntáronse o 15 de abril para considerar os posibles avances en enxeñaría xenética nos próximos cinco anos e calquera nova regulación que puidese necesitarse como resultado.[248] En 2017 a FDA propuxo unha regra pola que clasificarían as modificacións por enxeñaría xenética en animais como "fármacos animais", somenténdoos a estritas regulacións se se poñen á venda e reducindo a capacidade de individuos e pequenos negocios de sacarlles rendemento.[249][250]
Na China, onde as condicións sociais son moi diferentes das de Occidente, as enfermidades xenéticas teñen un forte estigma.[251] Isto levou á China a ter poucas barreiras regulatorias para o uso desta tecnoloxía.[252][253]
En 2012 e 2013, a tecnoloxía CRISPR foi un dos finalistas no premio ao descubrimento do ano de Science Magazine. En 2015 foi a vencedora do premio.[191] CRISPR foi considerado un dos 10 descubrimentos tecnolóxicos que elixe a revista MIT Technology Review en 2014 e 2016.[254][255] En 2016 Jennifer Doudna e Emmanuelle Charpentier, xunto con Rudolph Barrangou, Philippe Horvath e Feng Zhang gañaron o premio Gairdner International. En 2017 Doudna e Charpentier recibiron o Premio Xapón en Toquio pola súa invención revolucionaria do CRISPR-Cas9. En 2016, Charpentier, Doudna e Zhang gañaron o premio Tang de ciencias biofarmacéuticas.[256] En 2020 Charpentier e Doudna foron galardoadas co premio Nobel de Química, o primeiro que recibiu un equipo integramente feminino, "polo desenvolvemento dun método para a edición do xenoma."[257]