Мультиоміка
Мультиоміка, мульти-оміка, інтегративна оміка — це підхід до біологічного аналізу, спрямований на використання та інтеграцію великої кількості даних, наданої дослідженнями «-омами», такими як геном, протеом, транскриптом, епігеном, епітранскриптом[en], метаболом, інтерактом, мікробіом (метагеном, метатранскриптом, метапротеом) та інші, щоб розвинути комплексне та цілісне розуміння біологічних систем.[1][2][3][4]
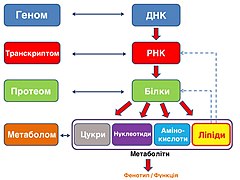

«Оміксні технології» — це набори передових, високопродуктивних методологій, які використовуються для аналізу широкого діапазону біологічних молекул та їх взаємодії з метою розуміння структури, функції та динаміки біологічних систем на різних рівнях.[5] Поєднуючи ці «-оми», вчені можуть аналізувати складні біологічні великі дані, щоб знаходити нові асоціації між біологічними об’єктами, точно визначати відповідні біомаркери[en] захворювань і фізіологічних процесів.[6][7] Роблячи це, мультиоміка об’єднує різноманітні дані омік, щоб знайти узгоджено відповідний зв’язок або асоціацію генотип-фенотип-довкілля.[8] Термін «мультіоміка» відображає взаємозв’язок і складну взаємодію між різними типами біологічних даних. Біологічні процеси керуються каскадом подій, які починаються з генома та призводять до функціональних білків і метаболітів, які опосередковують клітинні процеси. Уздовж цього континууму існують різні точки регулювання та зворотного зв’язку, які найкраще зрозуміти, якщо розглядати їх разом.[9]
Мультиоміка має на меті вийти за рамки редукціоністського погляду на розгляд кожного типу даних омік ізольовано. Натомість мультиоміка, як підхід, визнає, що ціле більше, ніж сума його частин. Підходи мультиоміки можуть прояснити, як зміни на одному рівні (наприклад, генетична мутація) можуть поширюватися на інші рівні (наприклад, зміни у функції білка та виробництві метаболітів), таким чином сприяючи складним рисам або хворобливим станам.[10][11]
Цей комплексний підхід є особливо ефективним у вирішенні складних біологічних питань і все частіше застосовується в таких сферах, як біомедицина[7] та біомедична інженерія, біотехнологія, експериментальна біологія, біоінженерія та синтетична біологія, сільське господарство й дослідження навколишнього середовища тощо. Наприклад, у персоналізованій медицині інтеграція мультиомних даних — від геномних до навколишніх — пропонує можливість більш точного прогнозування, профілактики та стратегій лікування для конкретних пацієнтів.[1][12][13][14][15]
Однак інтеграція кількох типів даних омік не позбавлена проблем. Вони включають потребу в надійних обчислювальних інструментах і статистичних методах для обробки та аналізу великих складних наборів даних; необхідність стандартизації та перевірки даних; а також питання етики, конфіденційності та обміну даними.[16] Незважаючи на ці проблеми, мультиоміка має величезні перспективи для трансформації нашого розуміння складних біологічних систем і хворобливих процесів, і розвивається експоненційно.[6]
Типи досліджень в мультиоміці


Зазвичай, дослідження мультиоміки поєднують певні оміксні технології, але ці дані також можуть бути поєднані і з іншими біомедичними даними.[1]
Геноміка
Геноміка — це наука про повний набір генів в організмі (геном), включаючи їх організацію, функції та взаємодію.[17] Завершення проекту «Геном людини» у 2003 році стало знаменним досягненням, яке проклало шлях для передових досліджень генетики людини та інших видів.[18] Методи в геноміці включають секвенування цілого генома[en][19][20], повногеномне дослідження асоціацій[en] (GWAS)[21] і порівняльну геноміку[en][22][23] та інші.[24][25] (див. Геноміка, Біоінформатика)
Епігеноміка
Епігеноміка — це дослідження повного набору епігенетичних модифікацій генетичного матеріалу клітини, відомого як епігеном.[26][27][28][29] Ці зміни можуть впливати на експресію генів, "вмикаючи" чи "вимикаючи" їх, не змінюючи базову послідовність ДНК.[30]
- Профіль метилювання ДНК: Вивчення статусу метилювання специфічних залишків цитозину в ДНК. Патерни метилювання часто асоціюються з глушінням генів і можуть бути проаналізовані за допомогою таких методів, як бісульфітне секвенування[en] та методи аналізу мікрочипів[en].[31]
- Дані модифікації гістонів — ChIP-Seq (Секвенування імунопреципітацією хроматину): визначення місць і моделей модифікацій гістонів (наприклад, ацетилювання, метилювання), пов’язаних з регуляцією генів. Секвенування ChIP поєднує імунопреципітацію хроматину з високопродуктивним секвенуванням.
- Секвенування малих РНК[en]: профілювання малих некодуючих РНК (наприклад, мікроРНК[32], малих інтерферуючих РНК), які відіграють вирішальну роль у регуляції генів після транскрипції.[33]
- Аналіз довгих некодуючих РНК (lncRNA): Дослідження функцій і регуляторних ролей довгих некодуючих РНК у експресії генів та епігенетичній регуляції.[34]
- Методи фіксації конформації хромосом — відображення тривимірної організації хроматину в клітинному ядрі. Дані Hi-C дають зрозуміти, як хроматинові петлі та взаємодії впливають на регуляцію генів і архітектуру геному.
- Повноепігеномне дослідження асоціацій[en] (EWAS) — Епігеномне профілювання метилювання ДНК: виявлення епігенетичних змін, пов’язаних із захворюваннями, ознаками та впливом навколишнього середовища, подібно до повногеномних досліджень асоціацій (GWAS) для генетичних варіацій.
Транскриптоміка
Транскриптоміка передбачає вивчення повного набору транскриптів РНК, які виробляються геномом за певних умов.[35] Такі методи, як мікрочипи та секвенування РНК[en] (RNA-seq), дозволяють дослідникам вимірювати зміни в експресії генів, виявляючи, як клітини реагують на різні стимули.[36] (Див. також Транскрипція (біологія))
Епітранскриптоміка
Епітранскриптоміка — це дослідження епітранскриптому[en][37][38] — функціонально відповідних модифікацій транскриптому, які можуть впливати на стабільність, локалізацію та трансляцію РНК.[39][29] Це поле дозволяє досліджувати інший рівень регуляції генів, який впливає на численні клітинні процеси та потенційно сприяє хворобливим станам.[40]
Епітранскриптоміка зосереджена на вивченні посттранскрипційних модифікацій молекул РНК. Ці модифікації, такі як N6-метиладенозин (m6A)[41][42] і 5-метилцитозин (m5C)[43][44][45], можуть впливати на стабільність, локалізацію та ефективність трансляції РНК.
Епітранскриптомне секвенування[en]: такі методи, як m6A-seq[46][47] і m5C-seq[48][49], використовуються для профілювання модифікацій РНК у транскриптомі. Ці дані допомагають ідентифікувати модифіковані ділянки РНК і зрозуміти їхню функціональну роль.
Протеоміка
Протеоміка передбачає широкомасштабне вивчення білків, включаючи їх структуру, функції та взаємодії, — протеом.[50][51][52][53][54] Мас-спектрометрія та білкові мікрочіпи є широко використовуваними методами в протеоміці.[55][56]
Функціональна протеоміка — дослідження білок-білкових взаємодій, субклітинної локалізації та функцій білка в клітинних шляхах.[57][58]
Ліпідоміка
Ліпідоміка — це широкомасштабне дослідження шляхів і мереж клітинних ліпідів у біологічних системах.[59] Ліпідоміка використовує методи аналітичної хімії та мас-спектрометрії[60] для ідентифікації та кількісного визначення різноманітного профілю ліпідів у біологічних системах.[61][62]
Глікоміка
Глікоміка — це комплексне дослідження всіх гліканових структур (вуглеводів) клітини, тканини або організму.[63] Глікоміка досліджує структуру та функції гліканів (цукрів, сахаридів) у біологічних системах, зокрема, завдяки мас-спектрометрії.[64][65][66]
Метаболоміка
Метаболоміка — це дослідження повного набору дрібномолекулярних хімічних речовин, знайдених у біологічному зразку. Досліджуючи ці метаболіти, дослідники можуть отримати уявлення про фізіологічні стани та реакцію на захворювання або зміни навколишнього середовища.[67][68] (див. Біоінформатика)
Інтерактоміка
Інтерактоміка відноситься до вивчення інтерактома, усієї сукупності молекулярних взаємодій у клітинах. Це може включати білок-білок[69][70], білок-ДНК[71], білок-метаболіт[72], епігенетичні[73], мікроРНК-мРНК[74], бактеріальні[75] та інші типи взаємодій.[76][77]
Мікробіоміка
Мікробіоміка передбачає характеристику та аналіз мікробних спільнот, включаючи бактерії, археї, віруси, гриби, найпростіші та водорості, у визначеному середовищі.[78] (Див. також Мікробіом, Мікробіом людини, Мікробіота кишківника)
Метагеноміка
Метагеноміка — це дослідження генетичного матеріалу, отриманого безпосередньо зі зразків навколишнього середовища[79] чи мікробіому певного організму.[80] Це дозволяє охарактеризувати спільноти організмів, які не піддаються культивуванню звичайними мікробіологічними методами.[81] Метагеноміка дає уявлення про структуру та функції мікробних спільнот у різноманітних середовищах, у тому числі в організмі людини.[82]
Метатранскриптоміка
Метатранскриптоміка — розділ транскриптоміки та мікробіоміки, який займається вивченням транскриптомів у мікробних спільнотах. Він пропонує розуміння активних функціональних елементів спільноти та може проілюструвати, як громади реагують на подразники навколишнього середовища. Метатранскриптоміка також може допомогти ідентифікувати гени, що експресуються окремими членами спільноти, досліджуючи функціональні ролі різних членів спільноти.[83][84]
Метапротеоміка
Метапротеоміка передбачає вивчення всього мікробного протеому в певний момент часу. Метапротеоміка надає функціональну інформацію про мікробні спільноти, що робить її ключовим інструментом для розуміння взаємодії мікробів і динаміки спільнот у їхньому природному середовищі існування. Такий підхід також допомагає з’ясувати функціональні ролі окремих членів і спільноти в цілому.[85][86][87]
Інструменти та методи
Секвенування наступного покоління
Технології секвенування наступного покоління (NGS) зробили революцію в геноміці та інших галузях оміки.[88] Вони забезпечують високопродуктивне секвенування, обробляючи мільйони послідовностей одночасно.[89][90] NGS використовується для секвенування цілих геномів, транскриптомів (RNA-seq), метагеномів і аналізу епігенетичних маркерів.[91][92][93][94]
Аналіз мікрочипів
Технологія мікрочипів[95], хоч і не така продуктивна як новіші методи, але також використовується для високопродуктивного аналізу експресії генів (транскриптоміка), а також для виявлення однонуклеотидних поліморфізмів (SNP) і варіацій кількості копій (CNV) у геноміці. Секвенування РНК (RNA-Seq), витіснило технологію мікрочіпів і стало методикою вибору, завдяки своїй здатності вимірювати експресію генів з високою чутливістю та точністю, а також ідентифікувати нові транскрипти.[96]
Мас-спектрометрія
Мас-спектрометрія є ключовим методом у протеоміці[56] та метаболоміці[68], що дозволяє ідентифікувати та кількісно визначити білки та метаболіти. Цей метод також використовують для ліпідоміки[60], глікоміки[65][66] та інших типів молекулярного аналізу.
Біоінформатика та обчислювальні засоби
Необхідним компонентом досліджень мультиоміки є використання обчислювальних інструментів для аналізу та інтеграції великих і складних наборів даних оміки, що є сферою досліджень в біоінформатиці. Інструменти для інтеграції даних включають статистичні підходи, алгоритми машинного навчання та інструменти мережевого аналізу.[97] Бази даних і програмні платформи[98][99] також мають вирішальне значення для обміну та анотування даних різних омік.[100]
Машинне навчання
Паралельно з прогресом у молекулярній, системній та обчислювальній біології, активно розвиваються програми машинного навчання для аналізу біомедичних даних. Інтеграція мультиомічного аналізу даних і машинного навчання призвела до відкриття нових біомаркерів.[101][102][103][104] Наприклад, один із методів проекту mixOmics реалізує метод, заснований на частковій регресії найменших квадратів[en] для відбору ознак (передбачуваних біомаркерів).[105]
Одноклітинна мультиоміка
Відгалуженням галузі мультиоміки є аналіз багаторівневих одноклітинних даних, які називають одноклітинною[en] мультиомікою.[106][107][108][109] Цей підхід дає нам безпрецедентну можливість розглядати багаторівневі процеси в здоров’ї та захворюваннях на рівні однієї клітини.[110]
Методи паралельного одноклітинного геномного та транскриптомного аналізу можуть базуватися на одночасній ампліфікації[111] або фізичному розділенні РНК і геномної ДНК.[112] Вони дозволяють отримати інформацію, яку неможливо зібрати виключно за допомогою транскриптомного аналізу, оскільки, наприклад, дані РНК не містять некодуючих геномних ділянок та інформації щодо варіації кількості копій. Розширенням цієї методології є інтеграція одноклітинних транскриптомів до одноклітинних метиломів, поєднання одноклітинного бісульфітного секвенування[en][113][114] до одноклітинного секвенування РНК (RNA-Seq).[115] Інші методи досліджують епігеном, такі як одноклітинний ATAC-seq[en][116] і одноклітинний Hi-C.[117]
Проблемою є інтеграція протеомних[51][54] і транскриптомних даних.[118][119] Один із підходів до виконання такого вимірювання полягає у фізичному розділенні одноклітинних лізатів на дві частини, обробці половини для РНК, а половини для білків.[118] Вміст білка в лізатах можна виміряти, наприклад, за допомогою методів розширення (PEA), які використовують антитіла зі штрих-кодом ДНК.[120] Інший підхід використовує комбінацію РНК-зондів важких металів і білкових антитіл для адаптації мас-цитометрії для мультиомічного аналізу.[119]
Системи CRISPR-Cas
Системи CRISPR-Cas, зокрема CRISPR-Cas9, зробили революцію в геномних дослідженнях. Їх можна використовувати для точного редагування геномів, що дозволяє проводити функціональні дослідження генів і генетичних варіантів.
Завдяки точності CRISPR-Cas9 дослідники можуть створювати певні генетичні пертубації (наприклад, генні нокаути або специфічні мутації) контрольованим чином, а потім використовувати різні методи оміки (геноміка, транскриптоміка, протеоміка, метаболоміка тощо), щоб зрозуміти наступні наслідки цих змін.[121] Це дозволяє отримати цілісне уявлення про вплив конкретного гена на процеси в клітині.
За тим же принципом, систему dCas9, де білок Cas9 є «мертвим» або каталітично неактивним, можна використовувати для цілеспрямованого редагування епігенома, якщо його злити з ферментами, які модифікують епігеном, такими як ДНК-метилтрансферази або гістон-ацетилтрансферази.[122] Після цього, методами епігеноміки можливо дослідити й оцінити отриманні зміни в епігеномі і їх роль в процесах в клітині.[123]
Інтеграція даних в мультиоміці
Інтеграція даних мультиоміки є вирішальним кроком у дослідженнях мультиоміки, метою якого є надання повного уявлення про біологічні системи на різних молекулярних рівнях. Завдання класифікації методів інтеграції даних мультиоміки є складним через різноманітні методології з різними цілями. Однак їх можна широко класифікувати на основі їхніх базових статистичних стратегій, їхньої біологічної цілі та способу обробки кількох типів даних омік.[4]
Існує два загальні підходи до організації вхідних даних для подальшої їх інтеграції та аналізу: методи ансамблю даних і методи ансамблю моделі. Методи ансамблю даних об’єднують дані мультиоміки з різних молекулярних шарів в єдину матрицю як вхідні дані. Навпаки, методи ансамблю моделі аналізують кожні дані омік незалежно, а потім об’єднують результати для побудови інтегративного аналізу.[124][125][126][127] На основі цих двох методик організації даних класифікують наступні методи інтеграції даних[126]:
Методи інтеграції даних
Методи на основі регресії/асоціації
- Послідовний аналіз
- Методи на основі канонічний кореляційного аналізу (CCA) і співінерційного аналізу (CIA)
- Методи на основі факторного аналізу
Методи на основі кластеризації
- Кластеризація на основі ядра
- Метод кластеризації на основі матричної факторизації
- Метод баєсової кластеризації
- Багатовимірний метод кластеризації
Методи на основі мереж
- Мережі на основі матриці факторизації (MF-Based)
- Баєсові мережі
- Мережі на основі поширення (NP-Based)
- Кореляційні та інші мережі
Проблеми та виклики
В інтеграції мультиомічних даних є кілька проблем та викликів які стоять перед дослідниками. Вони включають потребу в:
- Великих та високоякісні наборах даних, оскільки методи інтеграції часто вимагають великих обсягів даних для надійних результатів.
- Методи обробки відсутніх даних, оскільки не всі типи даних омік можуть бути доступними для всіх зразків.
- Ефективні методи нормалізації та трансформації для врахування відмінностей у масштабі та розподілі між різними типами даних омік.
- Методи врахування пакетниз ефектів та інших джерел небіологічної варіації.[128]
Оскільки дослідження мультиоміки стають все більш поширеними, виникає потреба в розробці більш складних методів інтеграції, програмних засобів[129], автоматизованих систем[130], технологічних процесів і баз даних. Очікується, що майбутні досягнення в галузі біоінформатики, машинного навчання та штучного інтелекту відіграватимуть вирішальну роль у цьому відношенні.[127][131]
Застосування
В медицині
Прогрес в мультиоміці обіцяє заповнити прогалини в розумінні людського здоров’я[1] та хвороб[132], і багато дослідників працюють над способами створення та аналізу даних, пов’язаних із хворобами. Перші застосування варіюються від розуміння взаємодій господар-патоген та інфекційних захворювань[133][134], онкопатологій[135], до кращого розуміння хронічних і складних неінфекційних захворювань[136], неврологічних патологій[137], і вдосконалення персоналізованої медицини[12][13][14][15].
Інтегруючи геномні, транскриптомні, протеомні та інші омічні дані, клініцисти можуть розробити всебічне розуміння здоров’я та хворобливого стану пацієнта, керуючи вибором найбільш ефективних методів лікування та профілактичних заходів. Наприклад, огляд 2023 року виділив такі 5 основних застосувань мультиоміки в трансляційній медицині: 1) виявлення молекулярних моделей, пов’язаних із захворюванням, 2) ідентифікація підтипу, 3) діагностика та прогноз, 4) прогнозування реакції на ліки та 5) розуміння регуляторних процесів.[124]
Розуміння механізмів захворювання
Інтегруючи кілька шарів оміки, дослідники можуть отримати глибше розуміння механізмів захворювання на молекулярному рівні. Наприклад, геномні дані можуть ідентифікувати генетичні варіанти, пов’язані з хворобою, транскриптомні дані можуть виявити пов’язані зміни в експресії генів, протеомні дані можуть показати зміни рівнів і модифікацій білка, а метаболомічні дані можуть ідентифікувати пов’язані метаболічні зміни.[124][138]
Класифікація захворювань
Дані мультиоміки можуть допомогти класифікувати та підтипувати захворювання, особливо складні захворювання, у більш конкретні категорії на основі їхніх молекулярних профілів. Це може покращити діагностику та лікування. Наприклад, різні підтипи раку або діабету можуть мати різні молекулярні профілі, які по-різному реагують на лікування.[139][124]
Виявлення біомаркерів
Дані мультиоміки можуть допомогти у відкритті біомаркерів для діагностики захворювань, прогнозу та відповіді на лікування. Шляхом порівняння профілів омік захворювання та здорового стану можна ідентифікувати потенційні біомаркери.[140][141]
Розробка ліків та прогнозування реакції на ліки
Мультиоміка також може відігравати вирішальну роль у відкритті та розробці ліків. Наприклад, розуміння омік-профілів захворювань може допомогти в ідентифікації потенційних мішеней ліків.[142][143][138] Крім того, дані мультиоміки можуть допомогти передбачити та зрозуміти реакцію на ліки.[144][145]
Комплексний проект мікробіома людини

Друга фаза проекту людського мікробіому вартістю 170 мільйонів доларів була зосереджена на інтеграції даних пацієнтів у різні набори даних омік, враховуючи генетику господаря, клінічну інформацію та склад мікробіому.[146][147] Перша фаза була зосереджена на характеристиці спільнот у різних місцях тіла. Друга фаза зосереджена на інтеграції мультиомічних даних від господаря та мікробіому до захворювань людини. Зокрема, проект використовував мультиоміку для покращення розуміння взаємодії мікробіот кишківника та носа з діабетом 2 типу[148], мікробіоти кишківника та запальних захворювань кишківника[149], та мікробіоти вагіни й передчасних пологів[150].
Системна імунологія
Складність взаємодій в імунній системі людини спонукала до створення великої кількості пов’язаних з імунологією багатомасштабних омічних даних.[151][152][153][13][154] Мультиомічний аналіз даних був використаний для збору нових ідей щодо імунної відповіді на інфекційні захворювання, такі як дитяча чікунгунья[155], а також неінфекційні аутоімунні захворювання.[156] Мультиоміку також активно використовували для розуміння ефективності та побічних ефектів вакцин.[157] Наприклад, мультиоміка була важливою для виявлення зв’язку змін метаболітів плазми та транскриптома імунної системи у відповідь на вакцинацію проти оперізувального герпесу.[158]
Старіння та омолодження
Старіння організму зумовлене взаємопов’язаними молекулярними змінами, що охоплюють внутрішні та позаклітинні фактори. Комбінований аналіз високопродуктивних наборів даних мультиоміки на популяційному або одноклітинному рівнях може забезпечити багатовимірний інтегрований профіль гетерогенних процесів старіння з безпрецедентною продуктивністю та деталізацією. Ці нові стратегії дозволяють досліджувати молекулярний профіль і регуляторний статус експресії генів під час старіння, і, у свою чергу, сприяють розробці нових заходів щодо старіння та омолодження.[159][160]
У дослідженні 2023 року, опублікованому в Nature Communications, дослідники використовували транскриптомні асоційовані дослідження (TWAS) і аналіз менделівської рандомізації (MR), щоб ідентифікувати сигнатури експресії генів і генетичні мішені ліків, пов’язані зі старінням на рівні епігеному та комплексним підходом до довголіття та омолодження (див. Механізми старіння). Вони ідентифікували 22 асоціації з високим ступенем достовірності з епігенетичним старінням і 7 з багатофакторним довголіттям, з кількома генами, пов’язаними з передачею сигналів інсуліну, функцією мітохондрій, клітинною реакцією на стрес і метаболізмом. Дослідження також виявило вплив імунних клітин на прискорення епігенетичного старіння, що передбачає потенційні можливі мішені для терапевтичних втручаннь. Подільші дослідження в цій сфері допоможуть визначити конкретні препарати чи втручання для терапії проти старіння. Інтеграція мультиоміки в точні стратегії омолодження є багатообіцяючою у визначенні лікарських цілей для продовження здорових років життя.[161]
В сільському господарстві
У галузі сільського господарства мультиомічні підходи застосовуються для покращення ефективності селекції[162] та генетичного поліпшення[163] сільськогосподарських культур і тварин. Геномні дані можуть ідентифікувати корисні генетичні ознаки, тоді як транскриптомні, протеомні та метаболомні дані можуть допомогти зрозуміти їх вплив. Це може керувати стратегіями розведення та зусиллями щодо генетичної модифікації.[164]
Також, застосування мультиоміки в сільському господарстві є важливим для сприяння сталим методам ведення сільського господарства. Оскільки надмірне використання хімічних добрив призвело до численних екологічних проблем, глибоке розуміння агроекосистеми, яка містить рослини, мікроби та ґрунт, є необхідним для сталого сільського господарства. Наприклад, мультиомічне дослідження структури агроекосистеми, опубліковане в PNAS в 2020 році, виявило, що органічний азот є ключовим компонентом, який сприяє врожайності сільськогосподарських культур за умови соляризації ґрунту, навіть при наявності неорганічного азоту.[165]
В екології
В екології та дослідженнях навколишнього середовища методи мультиоміки використовуються для вивчення екосистем на молекулярному рівні. Наприклад, метагеномні, метатранскриптомні, метапротеомні та метаметаболомні дані можуть дати розуміння складу та функції мікробних спільнот у різних середовищах та в конкретних ризосферах.[166][167][168][169]
Програмне забезпечення та бази даних
Проект Bioconductor курує різні пакети R, спрямовані на інтеграцію даних омік:
- omicade4 для множинного співінерційного аналізу мультиомічних наборів даних[170]
- MultiAssayExperiment, що пропонує інтерфейс біопровідника для накладання зразків[171]
- IMAS, пакет, зосереджений на використанні мультиомічних даних для оцінки альтернативного сплайсингу[172]
- bioCancer, пакет для візуалізації мультиомних даних раку[173]
- mixOmics, набір багатоваріантних методів для інтеграції даних[105]
- MultiDataSet, пакет для інкапсуляції кількох наборів даних[174]
База даних OmicTools[175] додаткововисвітлює R-пакети та інші інструменти для мультиомічного аналізу даних:
- PaintOmics, веб-ресурс для візуалізації наборів даних мультиоміки[176][177]
- SIGMA, програма Java, зосереджена на інтегрованому аналізі наборів даних раку[178]
- iOmicsPASS, інструмент на C++ для прогнозування фенотипу на основі мультиоміки[179]
- Grimon, графічний інтерфейс R для візуалізації мультиомічних даних[180]
- Omics Pipe, фреймворк на Python для відтворюваної автоматизації мультиомічного аналізу даних[181]
Мультиомні бази даних
Основним обмеженням класичних омічних досліджень є виділення лише одного рівня біологічної складності. Наприклад, транскриптомні дослідження можуть надавати інформацію на рівні транскриптів, але багато різних об’єктів та процесів впливають на біологічний стан зразка (геномні варіанти, посттрансляційні модифікації, продукти метаболізму, взаємодіючі організми тощо). З появою високопродуктивної біології стає все більш доступним проводити численні вимірювання, дозволяючи трансдоменні (наприклад, рівні РНК і білка) кореляції та висновки. Ці кореляції допомагають будувати більш повні біологічні мережі, заповнюючи прогалини в наших знаннях.
Інтеграція даних, однак, не є легким завданням. Щоб полегшити процес, були створені бази даних і конвеєри для систематичного дослідження мультиомічних даних:
- Multi-Omics Profiling Expression Database (MOPED)[182], що об’єднує різні моделі тварин
- База даних панкреатичної експресії, що об’єднує дані, пов’язані з тканиною підшлункової залози
- LinkedOmics[183][184] – об’єднання даних із наборів даних про рак TCGA
- OASIS[185] – веб-ресурс для загальних досліджень раку
- BCIP[186] – платформа для досліджень раку молочної залози
- C/VDdb[187] – поєднання даних кількох досліджень серцево-судинних захворювань
- ZikaVR[188] – мультиомічний ресурс для даних про вірус Зіка
- Ecomics[189] – нормалізована мультиомічна база даних для даних Escherichia coli
- GourdBase[190] – інтеграція даних із досліджень гарбуза
- MODEM[191] – база даних для багаторівневих даних про кукурудзу
- SoyKB[192] – база даних для багаторівневих даних про сою
- ProteomicsDB[193] – мультиоміка та багатоорганізмовий ресурс для наукових досліджень життя.
- Latch Registry[194] – інтегрована база даних для мультиоміксних досліджень.
Однак, бази даних містять різні представлення того самого біологічного шляху, що може призвести до різних результатів аналізу статистичного збагачення та прогнозних моделей у контексті точної медицини. Тому рекомендується використовувати бази даних декількох представлень шляхів або інтегративні бази даних.[195]
Див. також
Додаткова література
Книги
- Kang Ning (2023). Methodologies of Multi-Omics Data Integration and Data Mining: Techniques and Applications (англ). Сінгапур: Springer. с.167. ISBN 978-981-19-8209-5.
- Christopher Gerner, Michelle Hill (2021). Integrative Multi-Omics in Biomedical Research (англ, відкритий доступ). MDPI, Biomolecules. с.178. ISBN 978-3-0365-2582-2.
- Jong Hyuk Yoon, Chiara Villa (2021). Multi-Omics for the Understanding of Brain Diseases (англ., відкритий доступ). MDPI. 13 грудня 2021. ISBN 978-3-0365-2603-4.
Журнали
- Molecular Omics (Royal Society of Chemistry)
- OMICS: A Journal of Integrative Biology (Mary Ann Liebert Inc)
Статті
- Babu Mohan; Snyder Michael (червень 2023). Multi-Omics Profiling for Health. Molecular & Cellular Proteomics 22 (6). doi:10.1016/j.mcpro.2023.100561.
- Vandereyken Katy; Sifrim Alejandro; Thienpont Bernard; Voet Thierry (2 березня 2023). Methods and applications for single-cell and spatial multi-omics. Nature Reviews Genetics (англ.). с.1–22. doi:10.1038/s41576-023-00580-2.
- Athieniti Efi; Spyrou George M. (1 січня 2023). A guide to multi-omics data collection and integration for translational medicine. Computational and Structural Biotechnology Journal (англ.) 21. doi:10.1016/j.csbj.2022.11.050.
- Vahabi Nasim; Michailidis George (2022). Unsupervised Multi-Omics Data Integration Methods: A Comprehensive Review. Frontiers in Genetics 13. doi:10.3389/fgene.2022.854752.
- The Power of Multiomics. (2021). Illumina.
- Pinu Farhana R.; Beale David J.; Paten Amy M. та ін. (2019). Systems Biology and Multi-Omics Integration: Viewpoints from the Metabolomics Research Community. Metabolites, MDPI (англ.) 9 (4). doi:10.3390/metabo9040076.